Stockfotos & Bilder
(3,601)Stockfotos & Bilder
 Menschliche Gehirn und medizinisches Logo, Computer-Grafik. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-menschliche-gehirn-und-medizinisches-logo-computer-grafik-73688768.html
Menschliche Gehirn und medizinisches Logo, Computer-Grafik. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-menschliche-gehirn-und-medizinisches-logo-computer-grafik-73688768.htmlRFE7TPNM–Menschliche Gehirn und medizinisches Logo, Computer-Grafik.
 Aspirin Molekülmodell, Computer-Grafik. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-aspirin-molekulmodell-computer-grafik-73688767.html
Aspirin Molekülmodell, Computer-Grafik. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-aspirin-molekulmodell-computer-grafik-73688767.htmlRFE7TPNK–Aspirin Molekülmodell, Computer-Grafik.
 Proteinmoleküle, Computer-Grafik. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-proteinmolekule-computer-grafik-73688721.html
Proteinmoleküle, Computer-Grafik. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-proteinmolekule-computer-grafik-73688721.htmlRFE7TPM1–Proteinmoleküle, Computer-Grafik.
 Virus, Computer-Grafik. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-virus-computer-grafik-73689025.html
Virus, Computer-Grafik. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-virus-computer-grafik-73689025.htmlRFE7TR2W–Virus, Computer-Grafik.
 Proteinmoleküle, Computer-Grafik. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-proteinmolekule-computer-grafik-73688717.html
Proteinmoleküle, Computer-Grafik. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-proteinmolekule-computer-grafik-73688717.htmlRFE7TPKW–Proteinmoleküle, Computer-Grafik.
 Graphen, Computer-Grafik. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-graphen-computer-grafik-73688988.html
Graphen, Computer-Grafik. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-graphen-computer-grafik-73688988.htmlRFE7TR1G–Graphen, Computer-Grafik.
 Nanomodule, Computer-Grafik. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-nanomodule-computer-grafik-73689008.html
Nanomodule, Computer-Grafik. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-nanomodule-computer-grafik-73689008.htmlRFE7TR28–Nanomodule, Computer-Grafik.
 Menschliche Lunge, Computer-Grafik. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-menschliche-lunge-computer-grafik-73688998.html
Menschliche Lunge, Computer-Grafik. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-menschliche-lunge-computer-grafik-73688998.htmlRFE7TR1X–Menschliche Lunge, Computer-Grafik.
 Proteinmoleküle, Computer-Grafik. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-proteinmolekule-computer-grafik-73688726.html
Proteinmoleküle, Computer-Grafik. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-proteinmolekule-computer-grafik-73688726.htmlRFE7TPM6–Proteinmoleküle, Computer-Grafik.
 Nanomodules, Computer-Grafik. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-nanomodules-computer-grafik-73689006.html
Nanomodules, Computer-Grafik. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-nanomodules-computer-grafik-73689006.htmlRFE7TR26–Nanomodules, Computer-Grafik.
 H-Ras p21 Oncogene Protein, Molekülmodell. Die Ras-Proteine sind in der Übertragung von Signalen innerhalb der Zellen beteiligt. Übermäßige Signalisierung kann zu Erkrankungen wie Krebs führen, und dieses Protein ist daher als ein Onkogen Protein eingestuft. H-Ras (HRAS) ist auch kno Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-h-ras-p21-oncogene-protein-molekulmodell-die-ras-proteine-sind-in-der-ubertragung-von-signalen-innerhalb-der-zellen-beteiligt-ubermassige-signalisierung-kann-zu-erkrankungen-wie-krebs-fuhren-und-dieses-protein-ist-daher-als-ein-onkogen-protein-eingestuft-h-ras-hras-ist-auch-kno-73688579.html
H-Ras p21 Oncogene Protein, Molekülmodell. Die Ras-Proteine sind in der Übertragung von Signalen innerhalb der Zellen beteiligt. Übermäßige Signalisierung kann zu Erkrankungen wie Krebs führen, und dieses Protein ist daher als ein Onkogen Protein eingestuft. H-Ras (HRAS) ist auch kno Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-h-ras-p21-oncogene-protein-molekulmodell-die-ras-proteine-sind-in-der-ubertragung-von-signalen-innerhalb-der-zellen-beteiligt-ubermassige-signalisierung-kann-zu-erkrankungen-wie-krebs-fuhren-und-dieses-protein-ist-daher-als-ein-onkogen-protein-eingestuft-h-ras-hras-ist-auch-kno-73688579.htmlRFE7TPEY–H-Ras p21 Oncogene Protein, Molekülmodell. Die Ras-Proteine sind in der Übertragung von Signalen innerhalb der Zellen beteiligt. Übermäßige Signalisierung kann zu Erkrankungen wie Krebs führen, und dieses Protein ist daher als ein Onkogen Protein eingestuft. H-Ras (HRAS) ist auch kno
 Hepatitis-C-Glykoprotein und Antikörper. Molekulares Modell des E2-Umschlag-Glykoprotein aus dem Hepatitis C-Virus an eine neutralisierende Antikörper gebunden. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-hepatitis-c-glykoprotein-und-antikorper-molekulares-modell-des-e2-umschlag-glykoprotein-aus-dem-hepatitis-c-virus-an-eine-neutralisierende-antikorper-gebunden-73688370.html
Hepatitis-C-Glykoprotein und Antikörper. Molekulares Modell des E2-Umschlag-Glykoprotein aus dem Hepatitis C-Virus an eine neutralisierende Antikörper gebunden. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-hepatitis-c-glykoprotein-und-antikorper-molekulares-modell-des-e2-umschlag-glykoprotein-aus-dem-hepatitis-c-virus-an-eine-neutralisierende-antikorper-gebunden-73688370.htmlRFE7TP7E–Hepatitis-C-Glykoprotein und Antikörper. Molekulares Modell des E2-Umschlag-Glykoprotein aus dem Hepatitis C-Virus an eine neutralisierende Antikörper gebunden.
 H-Ras p21 Oncogene Protein, Molekülmodell. Die Ras-Proteine sind in der Übertragung von Signalen innerhalb der Zellen beteiligt. Übermäßige Signalisierung kann zu Erkrankungen wie Krebs führen, und dieses Protein ist daher als ein Onkogen Protein eingestuft. H-Ras (HRAS) ist auch kno Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-h-ras-p21-oncogene-protein-molekulmodell-die-ras-proteine-sind-in-der-ubertragung-von-signalen-innerhalb-der-zellen-beteiligt-ubermassige-signalisierung-kann-zu-erkrankungen-wie-krebs-fuhren-und-dieses-protein-ist-daher-als-ein-onkogen-protein-eingestuft-h-ras-hras-ist-auch-kno-73688576.html
H-Ras p21 Oncogene Protein, Molekülmodell. Die Ras-Proteine sind in der Übertragung von Signalen innerhalb der Zellen beteiligt. Übermäßige Signalisierung kann zu Erkrankungen wie Krebs führen, und dieses Protein ist daher als ein Onkogen Protein eingestuft. H-Ras (HRAS) ist auch kno Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-h-ras-p21-oncogene-protein-molekulmodell-die-ras-proteine-sind-in-der-ubertragung-von-signalen-innerhalb-der-zellen-beteiligt-ubermassige-signalisierung-kann-zu-erkrankungen-wie-krebs-fuhren-und-dieses-protein-ist-daher-als-ein-onkogen-protein-eingestuft-h-ras-hras-ist-auch-kno-73688576.htmlRFE7TPET–H-Ras p21 Oncogene Protein, Molekülmodell. Die Ras-Proteine sind in der Übertragung von Signalen innerhalb der Zellen beteiligt. Übermäßige Signalisierung kann zu Erkrankungen wie Krebs führen, und dieses Protein ist daher als ein Onkogen Protein eingestuft. H-Ras (HRAS) ist auch kno
 Xylose Isomerase Komplex. Molekulares Modell des Enzyms D-Xylose Isomerase verpflichtet, den Zucker Alkohol Sorbit. D-Xylose Isomerase ist Fruktose und Mannose-Stoffwechsel beteiligt. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-xylose-isomerase-komplex-molekulares-modell-des-enzyms-d-xylose-isomerase-verpflichtet-den-zucker-alkohol-sorbit-d-xylose-isomerase-ist-fruktose-und-mannose-stoffwechsel-beteiligt-73688578.html
Xylose Isomerase Komplex. Molekulares Modell des Enzyms D-Xylose Isomerase verpflichtet, den Zucker Alkohol Sorbit. D-Xylose Isomerase ist Fruktose und Mannose-Stoffwechsel beteiligt. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-xylose-isomerase-komplex-molekulares-modell-des-enzyms-d-xylose-isomerase-verpflichtet-den-zucker-alkohol-sorbit-d-xylose-isomerase-ist-fruktose-und-mannose-stoffwechsel-beteiligt-73688578.htmlRFE7TPEX–Xylose Isomerase Komplex. Molekulares Modell des Enzyms D-Xylose Isomerase verpflichtet, den Zucker Alkohol Sorbit. D-Xylose Isomerase ist Fruktose und Mannose-Stoffwechsel beteiligt.
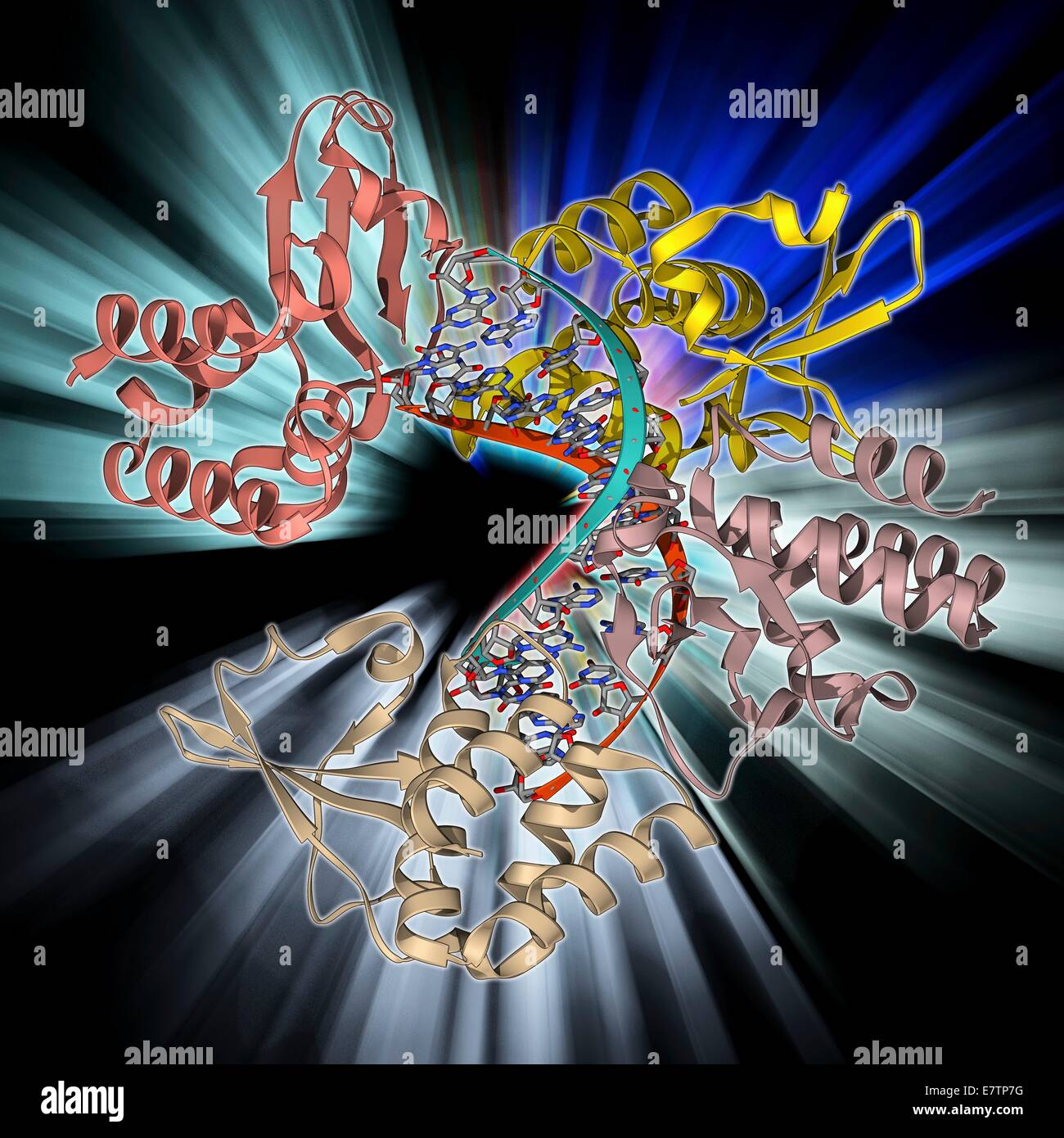 Marburg virales Protein 35 und RNA. Molekülmodell der Marburg virales Protein 35 (VP35) an ein Molekül von Double gebunden stranded RNA (Ribonukleinsäure). Dieses Protein hilft das Virus auf seinem Wirt Immunsystem zu entziehen. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-marburg-virales-protein-35-und-rna-molekulmodell-der-marburg-virales-protein-35-vp35-an-ein-molekul-von-double-gebunden-stranded-rna-ribonukleinsaure-dieses-protein-hilft-das-virus-auf-seinem-wirt-immunsystem-zu-entziehen-73688372.html
Marburg virales Protein 35 und RNA. Molekülmodell der Marburg virales Protein 35 (VP35) an ein Molekül von Double gebunden stranded RNA (Ribonukleinsäure). Dieses Protein hilft das Virus auf seinem Wirt Immunsystem zu entziehen. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-marburg-virales-protein-35-und-rna-molekulmodell-der-marburg-virales-protein-35-vp35-an-ein-molekul-von-double-gebunden-stranded-rna-ribonukleinsaure-dieses-protein-hilft-das-virus-auf-seinem-wirt-immunsystem-zu-entziehen-73688372.htmlRFE7TP7G–Marburg virales Protein 35 und RNA. Molekülmodell der Marburg virales Protein 35 (VP35) an ein Molekül von Double gebunden stranded RNA (Ribonukleinsäure). Dieses Protein hilft das Virus auf seinem Wirt Immunsystem zu entziehen.
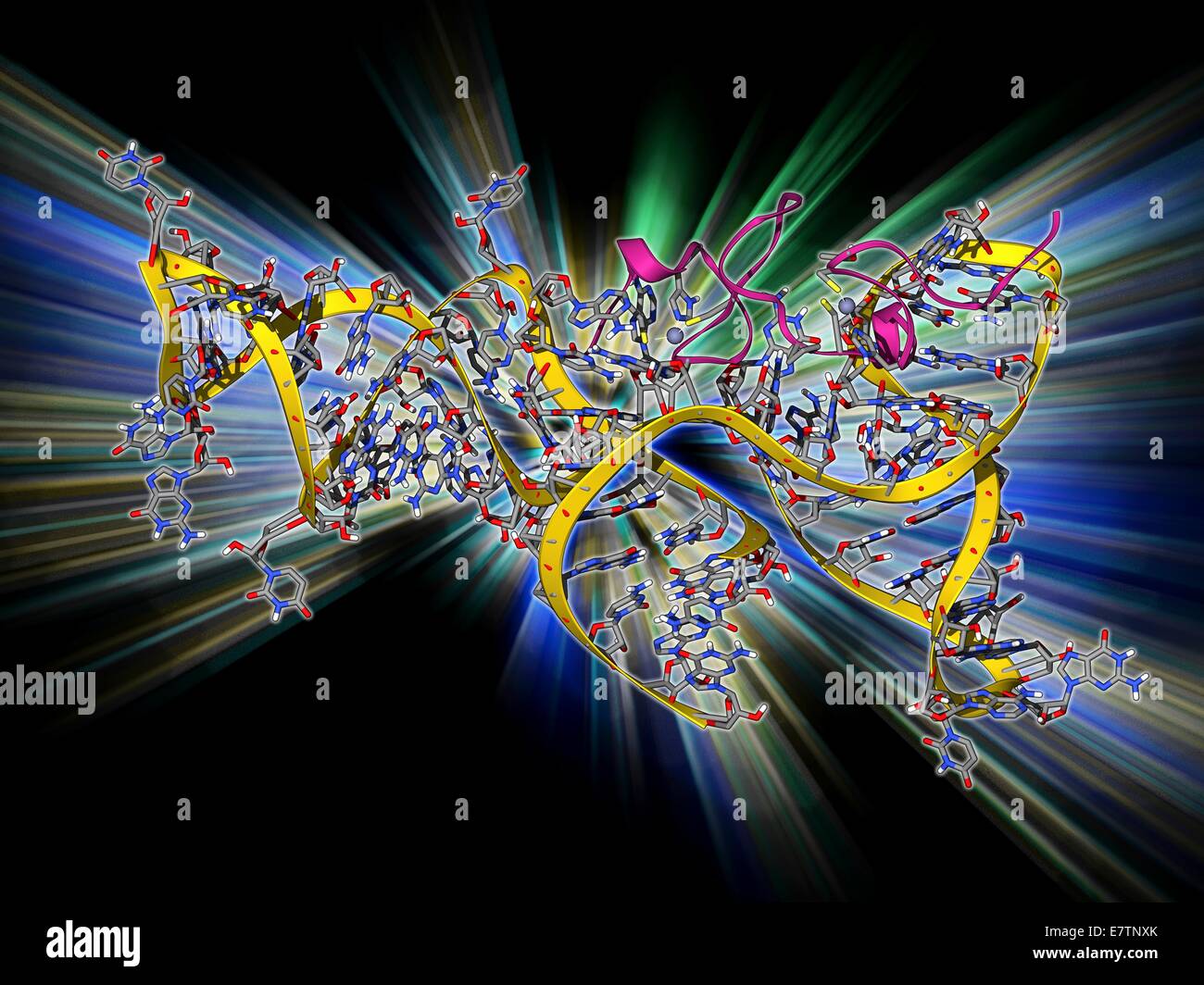 Virale RNA Verpackung Signal Komplex. Molekülmodell des MuPsi RNA Verpackung Signals Komplex von Rous-Sarkom-Vuris. Diese Sequenz RNA (Ribonukleinsäure) interagiert mit Nukleokapsid Domains, das Verpacken der RNA in Viruspartikel zu lenken. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-virale-rna-verpackung-signal-komplex-molekulmodell-des-mupsi-rna-verpackung-signals-komplex-von-rous-sarkom-vuris-diese-sequenz-rna-ribonukleinsaure-interagiert-mit-nukleokapsid-domains-das-verpacken-der-rna-in-viruspartikel-zu-lenken-73688123.html
Virale RNA Verpackung Signal Komplex. Molekülmodell des MuPsi RNA Verpackung Signals Komplex von Rous-Sarkom-Vuris. Diese Sequenz RNA (Ribonukleinsäure) interagiert mit Nukleokapsid Domains, das Verpacken der RNA in Viruspartikel zu lenken. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-virale-rna-verpackung-signal-komplex-molekulmodell-des-mupsi-rna-verpackung-signals-komplex-von-rous-sarkom-vuris-diese-sequenz-rna-ribonukleinsaure-interagiert-mit-nukleokapsid-domains-das-verpacken-der-rna-in-viruspartikel-zu-lenken-73688123.htmlRFE7TNXK–Virale RNA Verpackung Signal Komplex. Molekülmodell des MuPsi RNA Verpackung Signals Komplex von Rous-Sarkom-Vuris. Diese Sequenz RNA (Ribonukleinsäure) interagiert mit Nukleokapsid Domains, das Verpacken der RNA in Viruspartikel zu lenken.
 Fettsäure-bindendes Protein und Inhibitor. Molekulares Modell des Adipocyte Fettsäure-bindendes Protein (A-FABP) verpflichtet, ein Inhibitor. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-fettsaure-bindendes-protein-und-inhibitor-molekulares-modell-des-adipocyte-fettsaure-bindendes-protein-a-fabp-verpflichtet-ein-inhibitor-73688295.html
Fettsäure-bindendes Protein und Inhibitor. Molekulares Modell des Adipocyte Fettsäure-bindendes Protein (A-FABP) verpflichtet, ein Inhibitor. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-fettsaure-bindendes-protein-und-inhibitor-molekulares-modell-des-adipocyte-fettsaure-bindendes-protein-a-fabp-verpflichtet-ein-inhibitor-73688295.htmlRFE7TP4R–Fettsäure-bindendes Protein und Inhibitor. Molekulares Modell des Adipocyte Fettsäure-bindendes Protein (A-FABP) verpflichtet, ein Inhibitor.
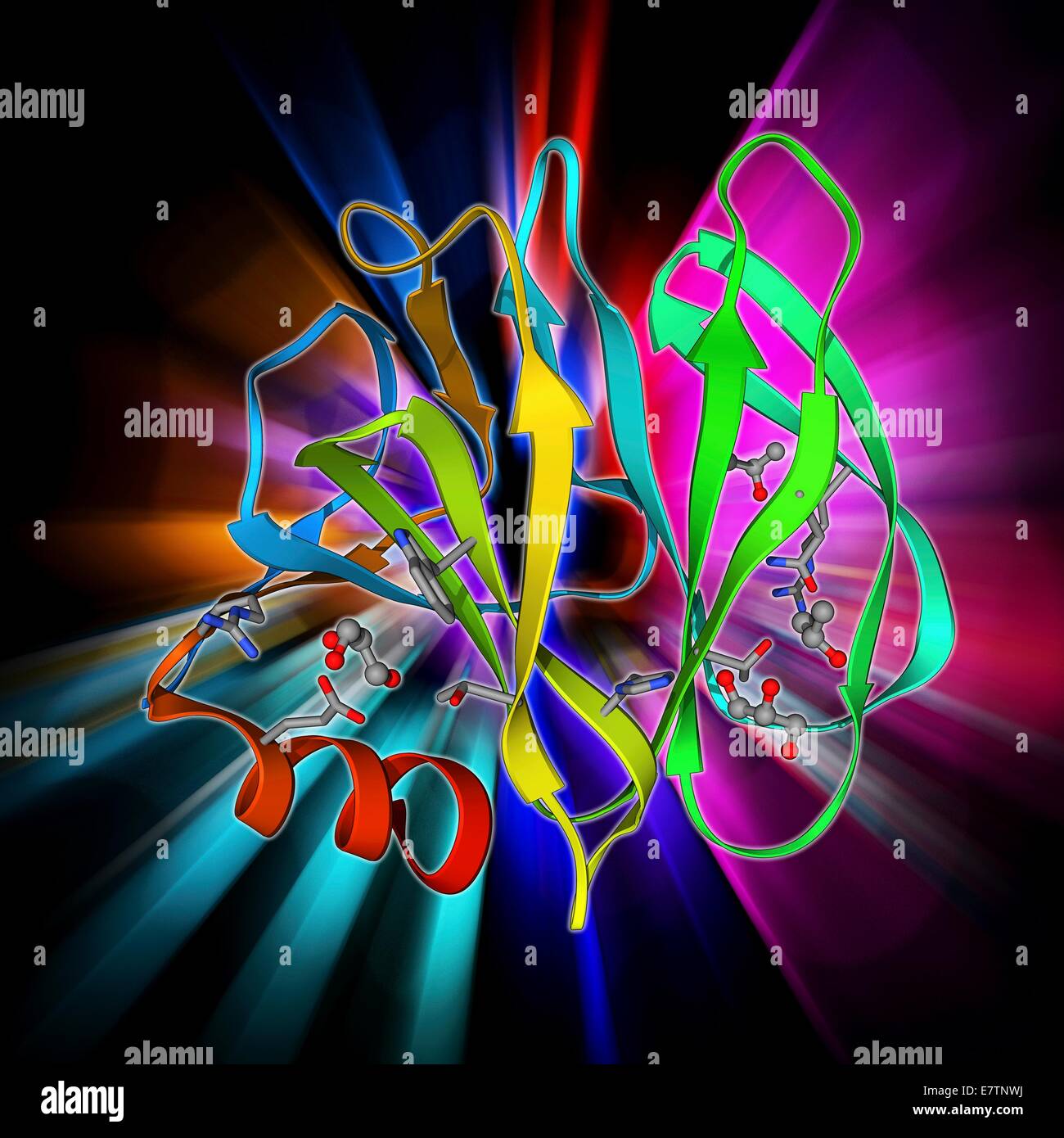 Menschliche Rotavirus Kapsid Protein, Molekülmodell. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-menschliche-rotavirus-kapsid-protein-molekulmodell-73688094.html
Menschliche Rotavirus Kapsid Protein, Molekülmodell. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-menschliche-rotavirus-kapsid-protein-molekulmodell-73688094.htmlRFE7TNWJ–Menschliche Rotavirus Kapsid Protein, Molekülmodell.
 Zinkfinger, Molekülmodell. Zinkfinger bilden die DNA Anerkennung Domänen von vielen DNA regulatorische Proteine und werden so für ihre Ähnlichkeit mit Fingern Projektion aus dem Protein genannt. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-zinkfinger-molekulmodell-zinkfinger-bilden-die-dna-anerkennung-domanen-von-vielen-dna-regulatorische-proteine-und-werden-so-fur-ihre-ahnlichkeit-mit-fingern-projektion-aus-dem-protein-genannt-73687969.html
Zinkfinger, Molekülmodell. Zinkfinger bilden die DNA Anerkennung Domänen von vielen DNA regulatorische Proteine und werden so für ihre Ähnlichkeit mit Fingern Projektion aus dem Protein genannt. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-zinkfinger-molekulmodell-zinkfinger-bilden-die-dna-anerkennung-domanen-von-vielen-dna-regulatorische-proteine-und-werden-so-fur-ihre-ahnlichkeit-mit-fingern-projektion-aus-dem-protein-genannt-73687969.htmlRFE7TNN5–Zinkfinger, Molekülmodell. Zinkfinger bilden die DNA Anerkennung Domänen von vielen DNA regulatorische Proteine und werden so für ihre Ähnlichkeit mit Fingern Projektion aus dem Protein genannt.
 Trypsinogen-Molekül. Molekulares Modell des Trypsinogen, der Vorläufer der verdauungsfördernden Protease Enzym Trypsin, komplexiert mit Inhibitor. Trypsin wird von der Bauchspeicheldrüse zum Abbau von Proteinen in kleinere Ketten von Aminosäuren freigesetzt. Es erscheint einer Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-trypsinogen-molekul-molekulares-modell-des-trypsinogen-der-vorlaufer-der-verdauungsfordernden-protease-enzym-trypsin-komplexiert-mit-inhibitor-trypsin-wird-von-der-bauchspeicheldruse-zum-abbau-von-proteinen-in-kleinere-ketten-von-aminosauren-freigesetzt-es-erscheint-einer-73687926.html
Trypsinogen-Molekül. Molekulares Modell des Trypsinogen, der Vorläufer der verdauungsfördernden Protease Enzym Trypsin, komplexiert mit Inhibitor. Trypsin wird von der Bauchspeicheldrüse zum Abbau von Proteinen in kleinere Ketten von Aminosäuren freigesetzt. Es erscheint einer Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-trypsinogen-molekul-molekulares-modell-des-trypsinogen-der-vorlaufer-der-verdauungsfordernden-protease-enzym-trypsin-komplexiert-mit-inhibitor-trypsin-wird-von-der-bauchspeicheldruse-zum-abbau-von-proteinen-in-kleinere-ketten-von-aminosauren-freigesetzt-es-erscheint-einer-73687926.htmlRFE7TNKJ–Trypsinogen-Molekül. Molekulares Modell des Trypsinogen, der Vorläufer der verdauungsfördernden Protease Enzym Trypsin, komplexiert mit Inhibitor. Trypsin wird von der Bauchspeicheldrüse zum Abbau von Proteinen in kleinere Ketten von Aminosäuren freigesetzt. Es erscheint einer
 Bakterielle Nanocompartment. Molekulare Molekül von einem Nanocompartment aus dem Bakterium Thermotoga Maritima. Dies ist eine Schale aus Enkapsulin Proteine gebildet, die Enzyme umschließt, die die Zellen vor oxidativem Stress zu schützen. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-bakterielle-nanocompartment-molekulare-molekul-von-einem-nanocompartment-aus-dem-bakterium-thermotoga-maritima-dies-ist-eine-schale-aus-enkapsulin-proteine-gebildet-die-enzyme-umschliesst-die-die-zellen-vor-oxidativem-stress-zu-schutzen-73688291.html
Bakterielle Nanocompartment. Molekulare Molekül von einem Nanocompartment aus dem Bakterium Thermotoga Maritima. Dies ist eine Schale aus Enkapsulin Proteine gebildet, die Enzyme umschließt, die die Zellen vor oxidativem Stress zu schützen. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-bakterielle-nanocompartment-molekulare-molekul-von-einem-nanocompartment-aus-dem-bakterium-thermotoga-maritima-dies-ist-eine-schale-aus-enkapsulin-proteine-gebildet-die-enzyme-umschliesst-die-die-zellen-vor-oxidativem-stress-zu-schutzen-73688291.htmlRFE7TP4K–Bakterielle Nanocompartment. Molekulare Molekül von einem Nanocompartment aus dem Bakterium Thermotoga Maritima. Dies ist eine Schale aus Enkapsulin Proteine gebildet, die Enzyme umschließt, die die Zellen vor oxidativem Stress zu schützen.
 Stickoxid-Synthase, Molekülmodell. Dieses Enzym katalysiert die Produktion von Stickoxid aus L-Arginin. Stickstoffmonoxid ist an zellulären Signalgebung beteiligt. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-stickoxid-synthase-molekulmodell-dieses-enzym-katalysiert-die-produktion-von-stickoxid-aus-l-arginin-stickstoffmonoxid-ist-an-zellularen-signalgebung-beteiligt-73687658.html
Stickoxid-Synthase, Molekülmodell. Dieses Enzym katalysiert die Produktion von Stickoxid aus L-Arginin. Stickstoffmonoxid ist an zellulären Signalgebung beteiligt. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-stickoxid-synthase-molekulmodell-dieses-enzym-katalysiert-die-produktion-von-stickoxid-aus-l-arginin-stickstoffmonoxid-ist-an-zellularen-signalgebung-beteiligt-73687658.htmlRFE7TNA2–Stickoxid-Synthase, Molekülmodell. Dieses Enzym katalysiert die Produktion von Stickoxid aus L-Arginin. Stickstoffmonoxid ist an zellulären Signalgebung beteiligt.
 Calcium-bindendes Protein. Molekül-Modell von Kalzium-bindende Protein Calmodulin (CaM). Dieses Protein findet sich in allen eukaryotischen Zellen, wo es reguliert und die Aktivitäten vieler Kalzium-bindende Enzyme ändert. Zelluläre Prozesse, dass CaM inc betrifft Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-calcium-bindendes-protein-molekul-modell-von-kalzium-bindende-protein-calmodulin-cam-dieses-protein-findet-sich-in-allen-eukaryotischen-zellen-wo-es-reguliert-und-die-aktivitaten-vieler-kalzium-bindende-enzyme-andert-zellulare-prozesse-dass-cam-inc-betrifft-73688286.html
Calcium-bindendes Protein. Molekül-Modell von Kalzium-bindende Protein Calmodulin (CaM). Dieses Protein findet sich in allen eukaryotischen Zellen, wo es reguliert und die Aktivitäten vieler Kalzium-bindende Enzyme ändert. Zelluläre Prozesse, dass CaM inc betrifft Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-calcium-bindendes-protein-molekul-modell-von-kalzium-bindende-protein-calmodulin-cam-dieses-protein-findet-sich-in-allen-eukaryotischen-zellen-wo-es-reguliert-und-die-aktivitaten-vieler-kalzium-bindende-enzyme-andert-zellulare-prozesse-dass-cam-inc-betrifft-73688286.htmlRFE7TP4E–Calcium-bindendes Protein. Molekül-Modell von Kalzium-bindende Protein Calmodulin (CaM). Dieses Protein findet sich in allen eukaryotischen Zellen, wo es reguliert und die Aktivitäten vieler Kalzium-bindende Enzyme ändert. Zelluläre Prozesse, dass CaM inc betrifft
 Nerve Growth Factor Rezeptor gebunden. Molekülmodell des Nerve Growth Factor (NGF) an den p75 Neurotrophin Rezeptor gebunden. NGF ist eine isolierte, die auf die Entwicklung und Funktion der Nerven wirkt. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-nerve-growth-factor-rezeptor-gebunden-molekulmodell-des-nerve-growth-factor-ngf-an-den-p75-neurotrophin-rezeptor-gebunden-ngf-ist-eine-isolierte-die-auf-die-entwicklung-und-funktion-der-nerven-wirkt-73687909.html
Nerve Growth Factor Rezeptor gebunden. Molekülmodell des Nerve Growth Factor (NGF) an den p75 Neurotrophin Rezeptor gebunden. NGF ist eine isolierte, die auf die Entwicklung und Funktion der Nerven wirkt. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-nerve-growth-factor-rezeptor-gebunden-molekulmodell-des-nerve-growth-factor-ngf-an-den-p75-neurotrophin-rezeptor-gebunden-ngf-ist-eine-isolierte-die-auf-die-entwicklung-und-funktion-der-nerven-wirkt-73687909.htmlRFE7TNK1–Nerve Growth Factor Rezeptor gebunden. Molekülmodell des Nerve Growth Factor (NGF) an den p75 Neurotrophin Rezeptor gebunden. NGF ist eine isolierte, die auf die Entwicklung und Funktion der Nerven wirkt.
 RNA Enzym, Molekülmodell bearbeiten. Dieses Enzym bindet an doppelsträngige RNA (Ribonukleinsäure). Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-rna-enzym-molekulmodell-bearbeiten-dieses-enzym-bindet-an-doppelstrangige-rna-ribonukleinsaure-73688129.html
RNA Enzym, Molekülmodell bearbeiten. Dieses Enzym bindet an doppelsträngige RNA (Ribonukleinsäure). Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-rna-enzym-molekulmodell-bearbeiten-dieses-enzym-bindet-an-doppelstrangige-rna-ribonukleinsaure-73688129.htmlRFE7TNXW–RNA Enzym, Molekülmodell bearbeiten. Dieses Enzym bindet an doppelsträngige RNA (Ribonukleinsäure).
 Importin Heterodimer Protein. Molekülmodell zeigt das importin Heterodimer, ein Komplex zwischen importin Alpha und Beta importin. Importin importiert andere Proteine in den Zellkern. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-importin-heterodimer-protein-molekulmodell-zeigt-das-importin-heterodimer-ein-komplex-zwischen-importin-alpha-und-beta-importin-importin-importiert-andere-proteine-in-den-zellkern-73687878.html
Importin Heterodimer Protein. Molekülmodell zeigt das importin Heterodimer, ein Komplex zwischen importin Alpha und Beta importin. Importin importiert andere Proteine in den Zellkern. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-importin-heterodimer-protein-molekulmodell-zeigt-das-importin-heterodimer-ein-komplex-zwischen-importin-alpha-und-beta-importin-importin-importiert-andere-proteine-in-den-zellkern-73687878.htmlRFE7TNHX–Importin Heterodimer Protein. Molekülmodell zeigt das importin Heterodimer, ein Komplex zwischen importin Alpha und Beta importin. Importin importiert andere Proteine in den Zellkern.
 Dynamin Enzym. Molekülmodell des Geschäftsfeldes Pleckstrin Homologie (PH) des Enzyms Dynamin. Domains sind strukturelle Regionen von Enzymen, die oft aktiv in biologischen Prozessen beteiligt sind. Dynamin ist in erster Linie verantwortlich für die Erleichterung der Querkräfte Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-dynamin-enzym-molekulmodell-des-geschaftsfeldes-pleckstrin-homologie-ph-des-enzyms-dynamin-domains-sind-strukturelle-regionen-von-enzymen-die-oft-aktiv-in-biologischen-prozessen-beteiligt-sind-dynamin-ist-in-erster-linie-verantwortlich-fur-die-erleichterung-der-querkrafte-73688095.html
Dynamin Enzym. Molekülmodell des Geschäftsfeldes Pleckstrin Homologie (PH) des Enzyms Dynamin. Domains sind strukturelle Regionen von Enzymen, die oft aktiv in biologischen Prozessen beteiligt sind. Dynamin ist in erster Linie verantwortlich für die Erleichterung der Querkräfte Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-dynamin-enzym-molekulmodell-des-geschaftsfeldes-pleckstrin-homologie-ph-des-enzyms-dynamin-domains-sind-strukturelle-regionen-von-enzymen-die-oft-aktiv-in-biologischen-prozessen-beteiligt-sind-dynamin-ist-in-erster-linie-verantwortlich-fur-die-erleichterung-der-querkrafte-73688095.htmlRFE7TNWK–Dynamin Enzym. Molekülmodell des Geschäftsfeldes Pleckstrin Homologie (PH) des Enzyms Dynamin. Domains sind strukturelle Regionen von Enzymen, die oft aktiv in biologischen Prozessen beteiligt sind. Dynamin ist in erster Linie verantwortlich für die Erleichterung der Querkräfte
 Cytochrom C Oxidase. Molekulares Modell eines Cytochrom C Oxidase Enzyms aus den Mitochondrien des Herzens eine Kuh. Cytochrom-Moleküle führen Oxidation und Reduktion Reaktionen für Elektronentransport, eine Kette von Reaktionen verwendet, um macht zelluläre Prozesse tha Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-cytochrom-c-oxidase-molekulares-modell-eines-cytochrom-c-oxidase-enzyms-aus-den-mitochondrien-des-herzens-eine-kuh-cytochrom-molekule-fuhren-oxidation-und-reduktion-reaktionen-fur-elektronentransport-eine-kette-von-reaktionen-verwendet-um-macht-zellulare-prozesse-tha-73687653.html
Cytochrom C Oxidase. Molekulares Modell eines Cytochrom C Oxidase Enzyms aus den Mitochondrien des Herzens eine Kuh. Cytochrom-Moleküle führen Oxidation und Reduktion Reaktionen für Elektronentransport, eine Kette von Reaktionen verwendet, um macht zelluläre Prozesse tha Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-cytochrom-c-oxidase-molekulares-modell-eines-cytochrom-c-oxidase-enzyms-aus-den-mitochondrien-des-herzens-eine-kuh-cytochrom-molekule-fuhren-oxidation-und-reduktion-reaktionen-fur-elektronentransport-eine-kette-von-reaktionen-verwendet-um-macht-zellulare-prozesse-tha-73687653.htmlRFE7TN9W–Cytochrom C Oxidase. Molekulares Modell eines Cytochrom C Oxidase Enzyms aus den Mitochondrien des Herzens eine Kuh. Cytochrom-Moleküle führen Oxidation und Reduktion Reaktionen für Elektronentransport, eine Kette von Reaktionen verwendet, um macht zelluläre Prozesse tha
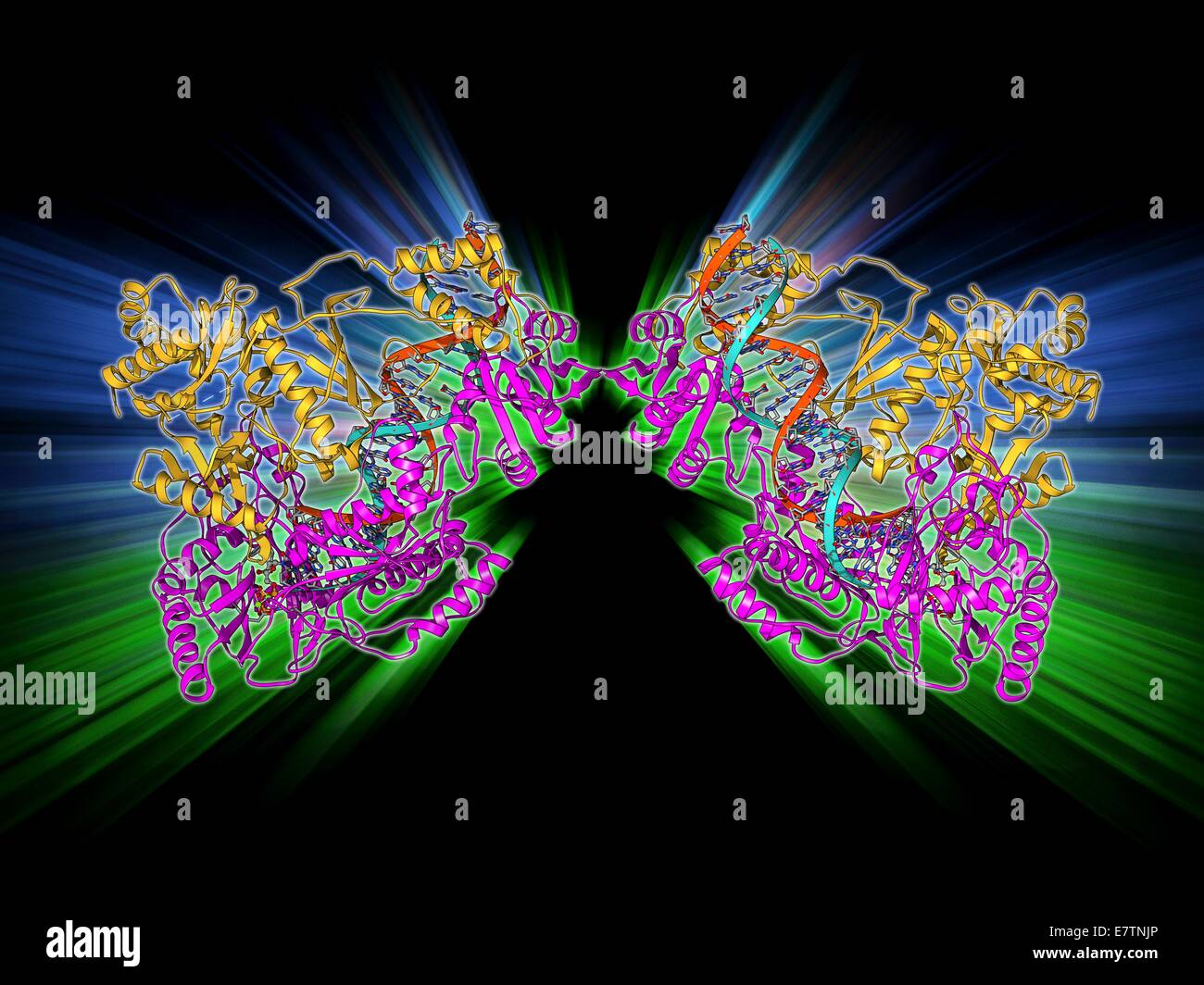 HIV-reverse Transkription Enzym. Molekülmodell des Enzyms Reverse Transkriptase (blau und grün) gefunden in HIV (Human Immunodeficiency Virus) komplexiert mit einem Molekül der DNA (Desoxyribonukleinsäure, rosa). Reverse Transkriptase überträgt die si Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-hiv-reverse-transkription-enzym-molekulmodell-des-enzyms-reverse-transkriptase-blau-und-grun-gefunden-in-hiv-human-immunodeficiency-virus-komplexiert-mit-einem-molekul-der-dna-desoxyribonukleinsaure-rosa-reverse-transkriptase-ubertragt-die-si-73687902.html
HIV-reverse Transkription Enzym. Molekülmodell des Enzyms Reverse Transkriptase (blau und grün) gefunden in HIV (Human Immunodeficiency Virus) komplexiert mit einem Molekül der DNA (Desoxyribonukleinsäure, rosa). Reverse Transkriptase überträgt die si Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-hiv-reverse-transkription-enzym-molekulmodell-des-enzyms-reverse-transkriptase-blau-und-grun-gefunden-in-hiv-human-immunodeficiency-virus-komplexiert-mit-einem-molekul-der-dna-desoxyribonukleinsaure-rosa-reverse-transkriptase-ubertragt-die-si-73687902.htmlRFE7TNJP–HIV-reverse Transkription Enzym. Molekülmodell des Enzyms Reverse Transkriptase (blau und grün) gefunden in HIV (Human Immunodeficiency Virus) komplexiert mit einem Molekül der DNA (Desoxyribonukleinsäure, rosa). Reverse Transkriptase überträgt die si
 Glykosylierung Enzym. Molekulares Modell des Enzyms N-Acetylglucosamin (GlcNAc) Transferase. Diese intrazelluläre Enzym verleiht N-Acetylglucosamin Moleküle Zielproteine. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-glykosylierung-enzym-molekulares-modell-des-enzyms-n-acetylglucosamin-glcnac-transferase-diese-intrazellulare-enzym-verleiht-n-acetylglucosamin-molekule-zielproteine-73687946.html
Glykosylierung Enzym. Molekulares Modell des Enzyms N-Acetylglucosamin (GlcNAc) Transferase. Diese intrazelluläre Enzym verleiht N-Acetylglucosamin Moleküle Zielproteine. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-glykosylierung-enzym-molekulares-modell-des-enzyms-n-acetylglucosamin-glcnac-transferase-diese-intrazellulare-enzym-verleiht-n-acetylglucosamin-molekule-zielproteine-73687946.htmlRFE7TNMA–Glykosylierung Enzym. Molekulares Modell des Enzyms N-Acetylglucosamin (GlcNAc) Transferase. Diese intrazelluläre Enzym verleiht N-Acetylglucosamin Moleküle Zielproteine.
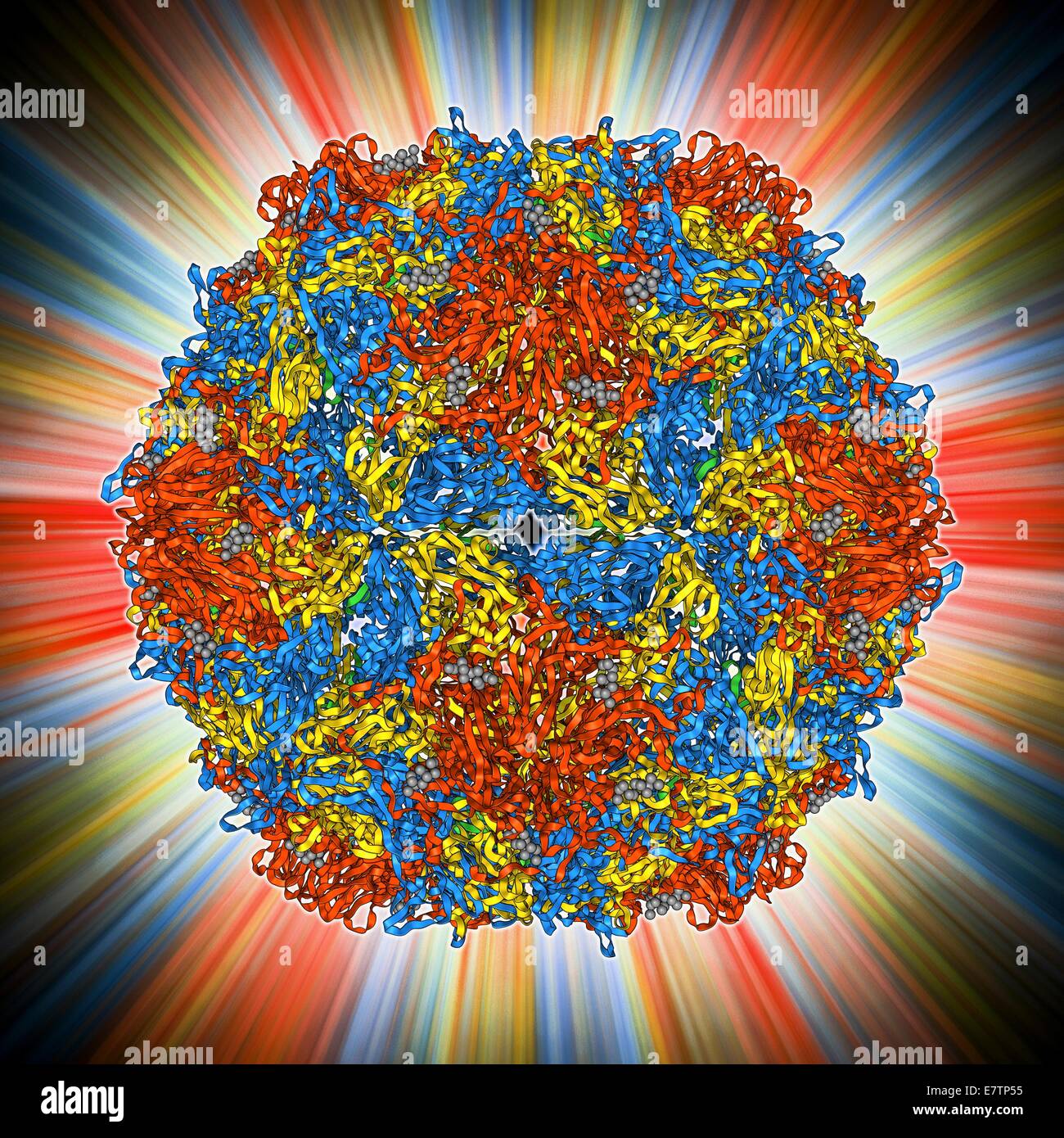 Poliovirus Typ 3 Kapsid, Molekülmodell. Diese Enteroviren verursacht Kinderlähmung (Polio) in den Menschen, die beeinflusst das Nervensystem, manchmal zu Lähmungen führt. Die drei Arten hervorrufen ähnliche Symptome. Viren wird das Kapsid die Protein-Shell-tha Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-poliovirus-typ-3-kapsid-molekulmodell-diese-enteroviren-verursacht-kinderlahmung-polio-in-den-menschen-die-beeinflusst-das-nervensystem-manchmal-zu-lahmungen-fuhrt-die-drei-arten-hervorrufen-ahnliche-symptome-viren-wird-das-kapsid-die-protein-shell-tha-73688305.html
Poliovirus Typ 3 Kapsid, Molekülmodell. Diese Enteroviren verursacht Kinderlähmung (Polio) in den Menschen, die beeinflusst das Nervensystem, manchmal zu Lähmungen führt. Die drei Arten hervorrufen ähnliche Symptome. Viren wird das Kapsid die Protein-Shell-tha Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-poliovirus-typ-3-kapsid-molekulmodell-diese-enteroviren-verursacht-kinderlahmung-polio-in-den-menschen-die-beeinflusst-das-nervensystem-manchmal-zu-lahmungen-fuhrt-die-drei-arten-hervorrufen-ahnliche-symptome-viren-wird-das-kapsid-die-protein-shell-tha-73688305.htmlRFE7TP55–Poliovirus Typ 3 Kapsid, Molekülmodell. Diese Enteroviren verursacht Kinderlähmung (Polio) in den Menschen, die beeinflusst das Nervensystem, manchmal zu Lähmungen führt. Die drei Arten hervorrufen ähnliche Symptome. Viren wird das Kapsid die Protein-Shell-tha
 E. Coli Virulenz Faktor Molekül. Molekulares Modell des Enzyms Arylsulfate Sulfotransferase (ASST) aus einem Bakterium Escherichia coli. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-e-coli-virulenz-faktor-molekul-molekulares-modell-des-enzyms-arylsulfate-sulfotransferase-asst-aus-einem-bakterium-escherichia-coli-73688294.html
E. Coli Virulenz Faktor Molekül. Molekulares Modell des Enzyms Arylsulfate Sulfotransferase (ASST) aus einem Bakterium Escherichia coli. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-e-coli-virulenz-faktor-molekul-molekulares-modell-des-enzyms-arylsulfate-sulfotransferase-asst-aus-einem-bakterium-escherichia-coli-73688294.htmlRFE7TP4P–E. Coli Virulenz Faktor Molekül. Molekulares Modell des Enzyms Arylsulfate Sulfotransferase (ASST) aus einem Bakterium Escherichia coli.
 Lysozym, Molekülmodell. Lysozymes sind Enzyme, die in einer Vielzahl von biologischen Flüssigkeiten wie Tränen, Speichel und Milch gefunden. Das Lysozym ist vom Huhn-Ei-weiß. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-lysozym-molekulmodell-lysozymes-sind-enzyme-die-in-einer-vielzahl-von-biologischen-flussigkeiten-wie-tranen-speichel-und-milch-gefunden-das-lysozym-ist-vom-huhn-ei-weiss-73688130.html
Lysozym, Molekülmodell. Lysozymes sind Enzyme, die in einer Vielzahl von biologischen Flüssigkeiten wie Tränen, Speichel und Milch gefunden. Das Lysozym ist vom Huhn-Ei-weiß. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-lysozym-molekulmodell-lysozymes-sind-enzyme-die-in-einer-vielzahl-von-biologischen-flussigkeiten-wie-tranen-speichel-und-milch-gefunden-das-lysozym-ist-vom-huhn-ei-weiss-73688130.htmlRFE7TNXX–Lysozym, Molekülmodell. Lysozymes sind Enzyme, die in einer Vielzahl von biologischen Flüssigkeiten wie Tränen, Speichel und Milch gefunden. Das Lysozym ist vom Huhn-Ei-weiß.
 Menschliche Rotavirus Enterotoxin. Molekülmodell der NSP4 (Elementoptionen Protein 4) von der menschlichen Rotaviren. Dieses Enterotoxin verursacht Durchfall. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-menschliche-rotavirus-enterotoxin-molekulmodell-der-nsp4-elementoptionen-protein-4-von-der-menschlichen-rotaviren-dieses-enterotoxin-verursacht-durchfall-73688320.html
Menschliche Rotavirus Enterotoxin. Molekülmodell der NSP4 (Elementoptionen Protein 4) von der menschlichen Rotaviren. Dieses Enterotoxin verursacht Durchfall. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-menschliche-rotavirus-enterotoxin-molekulmodell-der-nsp4-elementoptionen-protein-4-von-der-menschlichen-rotaviren-dieses-enterotoxin-verursacht-durchfall-73688320.htmlRFE7TP5M–Menschliche Rotavirus Enterotoxin. Molekülmodell der NSP4 (Elementoptionen Protein 4) von der menschlichen Rotaviren. Dieses Enterotoxin verursacht Durchfall.
 Rhinovirus Kapsid, Molekülmodell. Dies ist menschliche Rhinovirus. Das Rhinovirus befällt die obere Atemwege und ist die Ursache der Erkältung. Es wird durch Husten und Niesen verbreitet. Viren wird das Kapsid die Protein-Shell, die die genetische umschließt Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-rhinovirus-kapsid-molekulmodell-dies-ist-menschliche-rhinovirus-das-rhinovirus-befallt-die-obere-atemwege-und-ist-die-ursache-der-erkaltung-es-wird-durch-husten-und-niesen-verbreitet-viren-wird-das-kapsid-die-protein-shell-die-die-genetische-umschliesst-73688349.html
Rhinovirus Kapsid, Molekülmodell. Dies ist menschliche Rhinovirus. Das Rhinovirus befällt die obere Atemwege und ist die Ursache der Erkältung. Es wird durch Husten und Niesen verbreitet. Viren wird das Kapsid die Protein-Shell, die die genetische umschließt Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-rhinovirus-kapsid-molekulmodell-dies-ist-menschliche-rhinovirus-das-rhinovirus-befallt-die-obere-atemwege-und-ist-die-ursache-der-erkaltung-es-wird-durch-husten-und-niesen-verbreitet-viren-wird-das-kapsid-die-protein-shell-die-die-genetische-umschliesst-73688349.htmlRFE7TP6N–Rhinovirus Kapsid, Molekülmodell. Dies ist menschliche Rhinovirus. Das Rhinovirus befällt die obere Atemwege und ist die Ursache der Erkältung. Es wird durch Husten und Niesen verbreitet. Viren wird das Kapsid die Protein-Shell, die die genetische umschließt
 Parvovirus Partikel. Molekülmodell zeigt die Struktur der das Kapsid (äußere Protein Mantel) eines Partikels menschlicher Parvovirus (Familie Parvoviridae). Parvoviridae Viren sind die kleinsten bekannten Viren und einige der umweltfreundlichsten resistent. EAC Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-parvovirus-partikel-molekulmodell-zeigt-die-struktur-der-das-kapsid-aussere-protein-mantel-eines-partikels-menschlicher-parvovirus-familie-parvoviridae-parvoviridae-viren-sind-die-kleinsten-bekannten-viren-und-einige-der-umweltfreundlichsten-resistent-eac-73687907.html
Parvovirus Partikel. Molekülmodell zeigt die Struktur der das Kapsid (äußere Protein Mantel) eines Partikels menschlicher Parvovirus (Familie Parvoviridae). Parvoviridae Viren sind die kleinsten bekannten Viren und einige der umweltfreundlichsten resistent. EAC Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-parvovirus-partikel-molekulmodell-zeigt-die-struktur-der-das-kapsid-aussere-protein-mantel-eines-partikels-menschlicher-parvovirus-familie-parvoviridae-parvoviridae-viren-sind-die-kleinsten-bekannten-viren-und-einige-der-umweltfreundlichsten-resistent-eac-73687907.htmlRFE7TNJY–Parvovirus Partikel. Molekülmodell zeigt die Struktur der das Kapsid (äußere Protein Mantel) eines Partikels menschlicher Parvovirus (Familie Parvoviridae). Parvoviridae Viren sind die kleinsten bekannten Viren und einige der umweltfreundlichsten resistent. EAC
 Selenocystein Synthase Enzym-Molekül. Computer-Modell zeigt die molekulare Struktur des Enzyms Selenocystein Synthase (SecS). S O-Phospho-L-Seryl-tRNA wandelt Selenocysteyl-tRNA mit Selenophosphate als zusammengesetzte Selen Geber. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-selenocystein-synthase-enzym-molekul-computer-modell-zeigt-die-molekulare-struktur-des-enzyms-selenocystein-synthase-secs-s-o-phospho-l-seryl-trna-wandelt-selenocysteyl-trna-mit-selenophosphate-als-zusammengesetzte-selen-geber-73688279.html
Selenocystein Synthase Enzym-Molekül. Computer-Modell zeigt die molekulare Struktur des Enzyms Selenocystein Synthase (SecS). S O-Phospho-L-Seryl-tRNA wandelt Selenocysteyl-tRNA mit Selenophosphate als zusammengesetzte Selen Geber. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-selenocystein-synthase-enzym-molekul-computer-modell-zeigt-die-molekulare-struktur-des-enzyms-selenocystein-synthase-secs-s-o-phospho-l-seryl-trna-wandelt-selenocysteyl-trna-mit-selenophosphate-als-zusammengesetzte-selen-geber-73688279.htmlRFE7TP47–Selenocystein Synthase Enzym-Molekül. Computer-Modell zeigt die molekulare Struktur des Enzyms Selenocystein Synthase (SecS). S O-Phospho-L-Seryl-tRNA wandelt Selenocysteyl-tRNA mit Selenophosphate als zusammengesetzte Selen Geber.
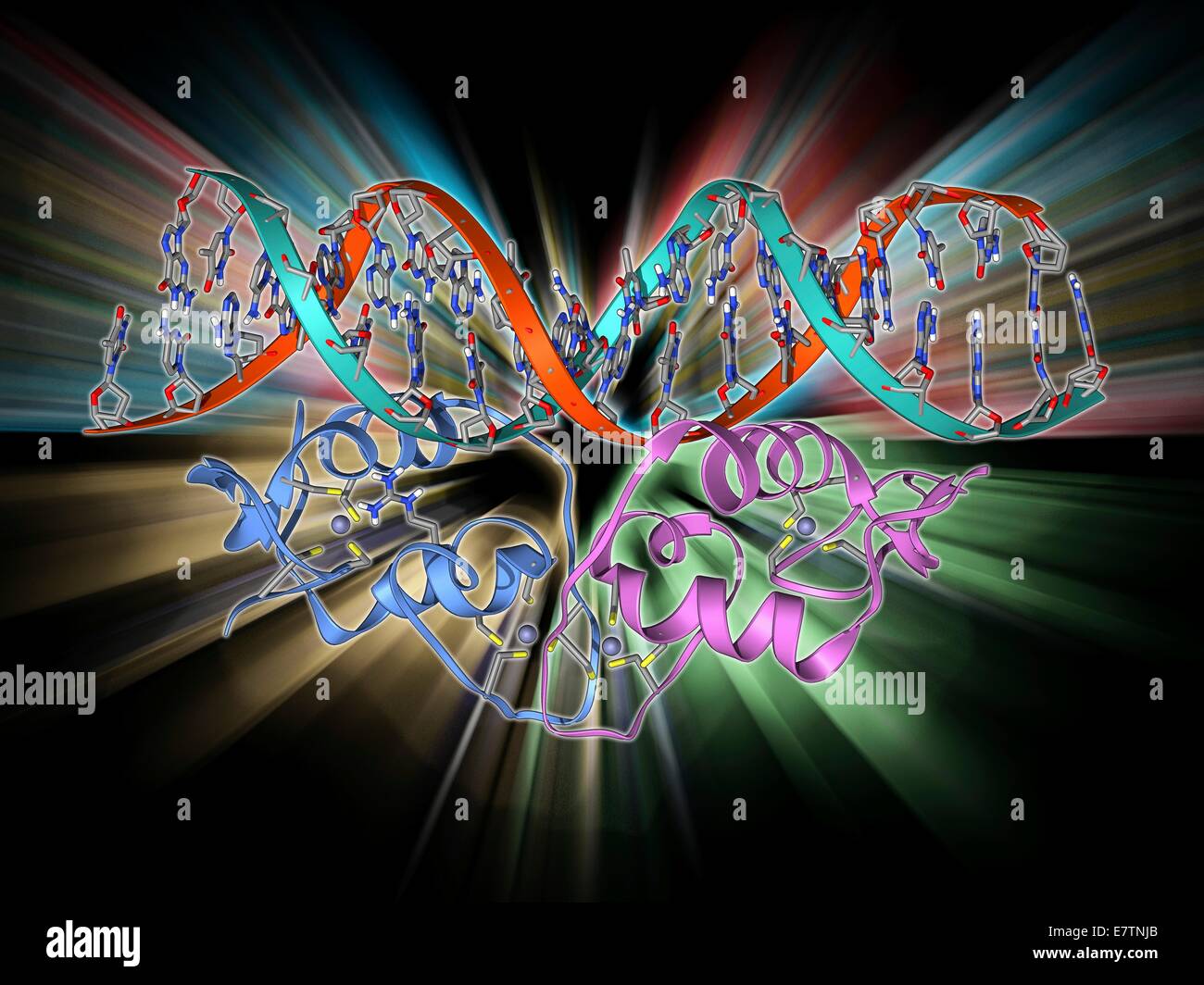 Transkriptionsfaktor und DNA-Molekül. Molekulares Modell des Glukokortikoid-Rezeptor (GR) Transkription Faktor Protein (Pink und blau) komplexiert mit einem Molekül der DNA (Desoxyribonukleinsäure, rot und blau). Transkriptionsfaktoren regulieren die Transkription des Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-transkriptionsfaktor-und-dna-molekul-molekulares-modell-des-glukokortikoid-rezeptor-gr-transkription-faktor-protein-pink-und-blau-komplexiert-mit-einem-molekul-der-dna-desoxyribonukleinsaure-rot-und-blau-transkriptionsfaktoren-regulieren-die-transkription-des-73687891.html
Transkriptionsfaktor und DNA-Molekül. Molekulares Modell des Glukokortikoid-Rezeptor (GR) Transkription Faktor Protein (Pink und blau) komplexiert mit einem Molekül der DNA (Desoxyribonukleinsäure, rot und blau). Transkriptionsfaktoren regulieren die Transkription des Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-transkriptionsfaktor-und-dna-molekul-molekulares-modell-des-glukokortikoid-rezeptor-gr-transkription-faktor-protein-pink-und-blau-komplexiert-mit-einem-molekul-der-dna-desoxyribonukleinsaure-rot-und-blau-transkriptionsfaktoren-regulieren-die-transkription-des-73687891.htmlRFE7TNJB–Transkriptionsfaktor und DNA-Molekül. Molekulares Modell des Glukokortikoid-Rezeptor (GR) Transkription Faktor Protein (Pink und blau) komplexiert mit einem Molekül der DNA (Desoxyribonukleinsäure, rot und blau). Transkriptionsfaktoren regulieren die Transkription des
 Parathion-Hydrolase, Molekülmodell. Dieses Enzym hydrolysiert Anleihen in Organophosphate, die Pestizide und das Nervengas Sarin enthalten. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-parathion-hydrolase-molekulmodell-dieses-enzym-hydrolysiert-anleihen-in-organophosphate-die-pestizide-und-das-nervengas-sarin-enthalten-73688342.html
Parathion-Hydrolase, Molekülmodell. Dieses Enzym hydrolysiert Anleihen in Organophosphate, die Pestizide und das Nervengas Sarin enthalten. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-parathion-hydrolase-molekulmodell-dieses-enzym-hydrolysiert-anleihen-in-organophosphate-die-pestizide-und-das-nervengas-sarin-enthalten-73688342.htmlRFE7TP6E–Parathion-Hydrolase, Molekülmodell. Dieses Enzym hydrolysiert Anleihen in Organophosphate, die Pestizide und das Nervengas Sarin enthalten.
 Dengue-Virus-Oberfläche Protein-Molekül. Molekulares Modell des das Umschlag-Glykoprotein auf der Oberfläche des Denguevirus hämorrhagisches Fieber (DHF) gefunden. Die Funktion dieses Proteins ist an der Oberfläche der Zielzelle zu binden und ermöglichen die virale Gene in Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-dengue-virus-oberflache-protein-molekul-molekulares-modell-des-das-umschlag-glykoprotein-auf-der-oberflache-des-denguevirus-hamorrhagisches-fieber-dhf-gefunden-die-funktion-dieses-proteins-ist-an-der-oberflache-der-zielzelle-zu-binden-und-ermoglichen-die-virale-gene-in-73687655.html
Dengue-Virus-Oberfläche Protein-Molekül. Molekulares Modell des das Umschlag-Glykoprotein auf der Oberfläche des Denguevirus hämorrhagisches Fieber (DHF) gefunden. Die Funktion dieses Proteins ist an der Oberfläche der Zielzelle zu binden und ermöglichen die virale Gene in Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-dengue-virus-oberflache-protein-molekul-molekulares-modell-des-das-umschlag-glykoprotein-auf-der-oberflache-des-denguevirus-hamorrhagisches-fieber-dhf-gefunden-die-funktion-dieses-proteins-ist-an-der-oberflache-der-zielzelle-zu-binden-und-ermoglichen-die-virale-gene-in-73687655.htmlRFE7TN9Y–Dengue-Virus-Oberfläche Protein-Molekül. Molekulares Modell des das Umschlag-Glykoprotein auf der Oberfläche des Denguevirus hämorrhagisches Fieber (DHF) gefunden. Die Funktion dieses Proteins ist an der Oberfläche der Zielzelle zu binden und ermöglichen die virale Gene in
 Nerve Growth Factor an Molekülmodell-Rezeptor gebunden. Menschlichen Nervenwachstumsfaktor gebunden an die TrkA Rezeptor. NGF ist eine isolierte, die auf die Entwicklung und Funktion der Nerven wirkt. TrkA bekannt auch als neurotrophen Tyrosin Kinase Rezeptor Typ 1. Th Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-nerve-growth-factor-an-molekulmodell-rezeptor-gebunden-menschlichen-nervenwachstumsfaktor-gebunden-an-die-trka-rezeptor-ngf-ist-eine-isolierte-die-auf-die-entwicklung-und-funktion-der-nerven-wirkt-trka-bekannt-auch-als-neurotrophen-tyrosin-kinase-rezeptor-typ-1-th-73687952.html
Nerve Growth Factor an Molekülmodell-Rezeptor gebunden. Menschlichen Nervenwachstumsfaktor gebunden an die TrkA Rezeptor. NGF ist eine isolierte, die auf die Entwicklung und Funktion der Nerven wirkt. TrkA bekannt auch als neurotrophen Tyrosin Kinase Rezeptor Typ 1. Th Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-nerve-growth-factor-an-molekulmodell-rezeptor-gebunden-menschlichen-nervenwachstumsfaktor-gebunden-an-die-trka-rezeptor-ngf-ist-eine-isolierte-die-auf-die-entwicklung-und-funktion-der-nerven-wirkt-trka-bekannt-auch-als-neurotrophen-tyrosin-kinase-rezeptor-typ-1-th-73687952.htmlRFE7TNMG–Nerve Growth Factor an Molekülmodell-Rezeptor gebunden. Menschlichen Nervenwachstumsfaktor gebunden an die TrkA Rezeptor. NGF ist eine isolierte, die auf die Entwicklung und Funktion der Nerven wirkt. TrkA bekannt auch als neurotrophen Tyrosin Kinase Rezeptor Typ 1. Th
 Eisen-regulatorischen Protein. Molekulares Modell des Eisen regulatorischen Protein 1 (IRP1). Abhängigkeit von der Konformation von IRP1 es können Handlungen als Regulator der mRNA (Messenger RNA) oder ein Enzym. IRP1 spielt eine Schlüsselrolle bei der Regulation des menschlichen Eisenstoffwechsels. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-eisen-regulatorischen-protein-molekulares-modell-des-eisen-regulatorischen-protein-1-irp1-abhangigkeit-von-der-konformation-von-irp1-es-konnen-handlungen-als-regulator-der-mrna-messenger-rna-oder-ein-enzym-irp1-spielt-eine-schlusselrolle-bei-der-regulation-des-menschlichen-eisenstoffwechsels-73688077.html
Eisen-regulatorischen Protein. Molekulares Modell des Eisen regulatorischen Protein 1 (IRP1). Abhängigkeit von der Konformation von IRP1 es können Handlungen als Regulator der mRNA (Messenger RNA) oder ein Enzym. IRP1 spielt eine Schlüsselrolle bei der Regulation des menschlichen Eisenstoffwechsels. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-eisen-regulatorischen-protein-molekulares-modell-des-eisen-regulatorischen-protein-1-irp1-abhangigkeit-von-der-konformation-von-irp1-es-konnen-handlungen-als-regulator-der-mrna-messenger-rna-oder-ein-enzym-irp1-spielt-eine-schlusselrolle-bei-der-regulation-des-menschlichen-eisenstoffwechsels-73688077.htmlRFE7TNW1–Eisen-regulatorischen Protein. Molekulares Modell des Eisen regulatorischen Protein 1 (IRP1). Abhängigkeit von der Konformation von IRP1 es können Handlungen als Regulator der mRNA (Messenger RNA) oder ein Enzym. IRP1 spielt eine Schlüsselrolle bei der Regulation des menschlichen Eisenstoffwechsels.
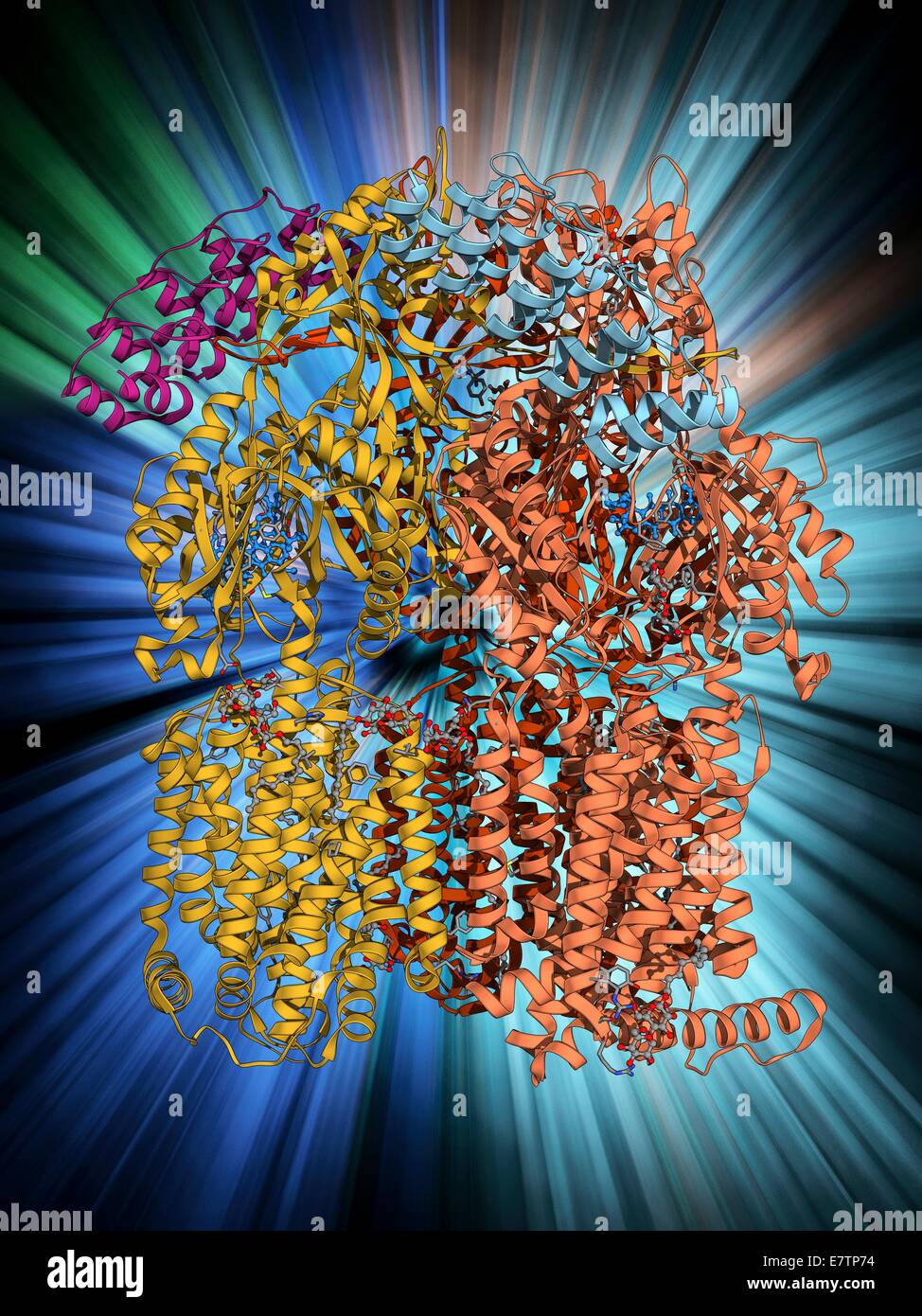 Multidrug Efflux Pumpe. Molekülmodell der multidrug Efflux Pumpe AcrB aus dem Bakterium Escherichia coli zwei Doxorubicin Moleküle zu transportieren. Dieses Protein Pumpen Drogen, einschließlich Antibiotika, aus der Bakterienzelle. Doxorubicin ist eine Chemotherapie Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-multidrug-efflux-pumpe-molekulmodell-der-multidrug-efflux-pumpe-acrb-aus-dem-bakterium-escherichia-coli-zwei-doxorubicin-molekule-zu-transportieren-dieses-protein-pumpen-drogen-einschliesslich-antibiotika-aus-der-bakterienzelle-doxorubicin-ist-eine-chemotherapie-73688360.html
Multidrug Efflux Pumpe. Molekülmodell der multidrug Efflux Pumpe AcrB aus dem Bakterium Escherichia coli zwei Doxorubicin Moleküle zu transportieren. Dieses Protein Pumpen Drogen, einschließlich Antibiotika, aus der Bakterienzelle. Doxorubicin ist eine Chemotherapie Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-multidrug-efflux-pumpe-molekulmodell-der-multidrug-efflux-pumpe-acrb-aus-dem-bakterium-escherichia-coli-zwei-doxorubicin-molekule-zu-transportieren-dieses-protein-pumpen-drogen-einschliesslich-antibiotika-aus-der-bakterienzelle-doxorubicin-ist-eine-chemotherapie-73688360.htmlRFE7TP74–Multidrug Efflux Pumpe. Molekülmodell der multidrug Efflux Pumpe AcrB aus dem Bakterium Escherichia coli zwei Doxorubicin Moleküle zu transportieren. Dieses Protein Pumpen Drogen, einschließlich Antibiotika, aus der Bakterienzelle. Doxorubicin ist eine Chemotherapie
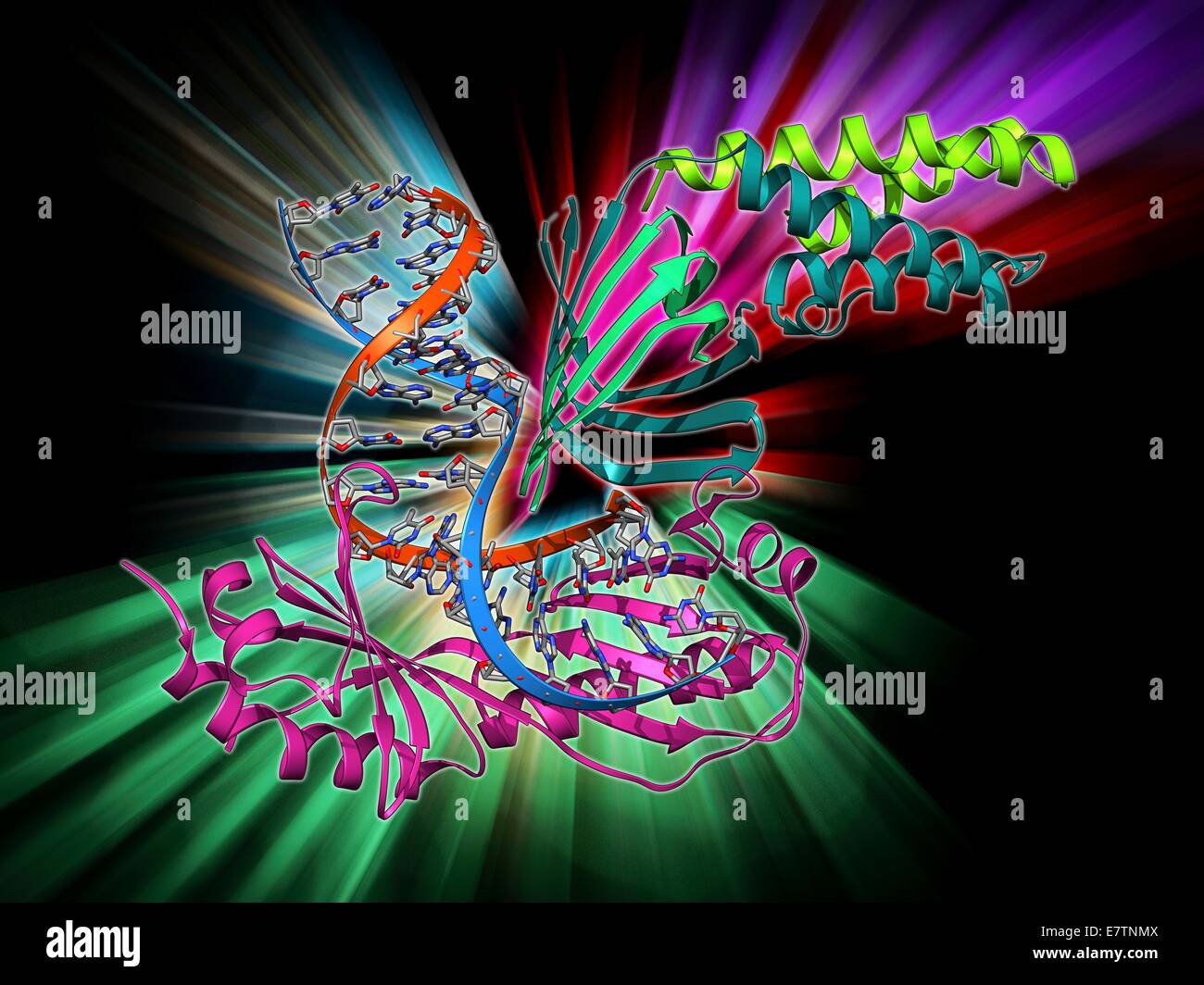 TATA-Box-bindendes Protein Komplex. Molekülmodell zeigt eine Hefe TATA-Box-bindendes Protein (TBP) komplexiert mit einem Strang der DNA (Desoxyribonukleinsäure, rot und blau) und Transkriptionsfaktor IIA. TBP ist eine allgemeine Transkriptionsfaktor, der umweltbedingten bindet Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-tata-box-bindendes-protein-komplex-molekulmodell-zeigt-eine-hefe-tata-box-bindendes-protein-tbp-komplexiert-mit-einem-strang-der-dna-desoxyribonukleinsaure-rot-und-blau-und-transkriptionsfaktor-iia-tbp-ist-eine-allgemeine-transkriptionsfaktor-der-umweltbedingten-bindet-73687962.html
TATA-Box-bindendes Protein Komplex. Molekülmodell zeigt eine Hefe TATA-Box-bindendes Protein (TBP) komplexiert mit einem Strang der DNA (Desoxyribonukleinsäure, rot und blau) und Transkriptionsfaktor IIA. TBP ist eine allgemeine Transkriptionsfaktor, der umweltbedingten bindet Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-tata-box-bindendes-protein-komplex-molekulmodell-zeigt-eine-hefe-tata-box-bindendes-protein-tbp-komplexiert-mit-einem-strang-der-dna-desoxyribonukleinsaure-rot-und-blau-und-transkriptionsfaktor-iia-tbp-ist-eine-allgemeine-transkriptionsfaktor-der-umweltbedingten-bindet-73687962.htmlRFE7TNMX–TATA-Box-bindendes Protein Komplex. Molekülmodell zeigt eine Hefe TATA-Box-bindendes Protein (TBP) komplexiert mit einem Strang der DNA (Desoxyribonukleinsäure, rot und blau) und Transkriptionsfaktor IIA. TBP ist eine allgemeine Transkriptionsfaktor, der umweltbedingten bindet
 Cytochrom BC1, Molekülmodell. Cytochrom-Moleküle führen Oxidation und Reduktion Reaktionen für Elektronentransport, eine Kette von Reaktionen verwendet, um macht zelluläre Prozesse, die Energie benötigen. Wie Zellfarbstoffe führen ein wichtiger Schritt bei der Herstellung von energ Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-cytochrom-bc1-molekulmodell-cytochrom-molekule-fuhren-oxidation-und-reduktion-reaktionen-fur-elektronentransport-eine-kette-von-reaktionen-verwendet-um-macht-zellulare-prozesse-die-energie-benotigen-wie-zellfarbstoffe-fuhren-ein-wichtiger-schritt-bei-der-herstellung-von-energ-73688300.html
Cytochrom BC1, Molekülmodell. Cytochrom-Moleküle führen Oxidation und Reduktion Reaktionen für Elektronentransport, eine Kette von Reaktionen verwendet, um macht zelluläre Prozesse, die Energie benötigen. Wie Zellfarbstoffe führen ein wichtiger Schritt bei der Herstellung von energ Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-cytochrom-bc1-molekulmodell-cytochrom-molekule-fuhren-oxidation-und-reduktion-reaktionen-fur-elektronentransport-eine-kette-von-reaktionen-verwendet-um-macht-zellulare-prozesse-die-energie-benotigen-wie-zellfarbstoffe-fuhren-ein-wichtiger-schritt-bei-der-herstellung-von-energ-73688300.htmlRFE7TP50–Cytochrom BC1, Molekülmodell. Cytochrom-Moleküle führen Oxidation und Reduktion Reaktionen für Elektronentransport, eine Kette von Reaktionen verwendet, um macht zelluläre Prozesse, die Energie benötigen. Wie Zellfarbstoffe führen ein wichtiger Schritt bei der Herstellung von energ
 Pilz Prion-Proteins. Molekulares Modell des Amyloid Form des Prion-Proteins HET. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-pilz-prion-proteins-molekulares-modell-des-amyloid-form-des-prion-proteins-het-73688161.html
Pilz Prion-Proteins. Molekulares Modell des Amyloid Form des Prion-Proteins HET. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-pilz-prion-proteins-molekulares-modell-des-amyloid-form-des-prion-proteins-het-73688161.htmlRFE7TP01–Pilz Prion-Proteins. Molekulares Modell des Amyloid Form des Prion-Proteins HET.
 Isocitrate Dehydrogenase, Molekülmodell. Dieses Enzym katalysiert die dritte Stufe der Zitronensäure (oder Krebs) Zyklus, den Prozess durch die Mitochondrien Glukose in Energie umwandeln. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-isocitrate-dehydrogenase-molekulmodell-dieses-enzym-katalysiert-die-dritte-stufe-der-zitronensaure-oder-krebs-zyklus-den-prozess-durch-die-mitochondrien-glukose-in-energie-umwandeln-73688281.html
Isocitrate Dehydrogenase, Molekülmodell. Dieses Enzym katalysiert die dritte Stufe der Zitronensäure (oder Krebs) Zyklus, den Prozess durch die Mitochondrien Glukose in Energie umwandeln. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-isocitrate-dehydrogenase-molekulmodell-dieses-enzym-katalysiert-die-dritte-stufe-der-zitronensaure-oder-krebs-zyklus-den-prozess-durch-die-mitochondrien-glukose-in-energie-umwandeln-73688281.htmlRFE7TP49–Isocitrate Dehydrogenase, Molekülmodell. Dieses Enzym katalysiert die dritte Stufe der Zitronensäure (oder Krebs) Zyklus, den Prozess durch die Mitochondrien Glukose in Energie umwandeln.
 Natrium-Kalium-Ionen Pumpe Protein, Molekülmodell. Natrium-Kalium-ATPase (Adenosin Triphosphatase) ist ein ATP-powered Ion Pumpe in allen tierischen Zellen gefunden. Es ist verantwortlich für die Aufrechterhaltung der hohen Konzentration von Kalium-Ionen und niedrige Konzentration o Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-natrium-kalium-ionen-pumpe-protein-molekulmodell-natrium-kalium-atpase-adenosin-triphosphatase-ist-ein-atp-powered-ion-pumpe-in-allen-tierischen-zellen-gefunden-es-ist-verantwortlich-fur-die-aufrechterhaltung-der-hohen-konzentration-von-kalium-ionen-und-niedrige-konzentration-o-73688275.html
Natrium-Kalium-Ionen Pumpe Protein, Molekülmodell. Natrium-Kalium-ATPase (Adenosin Triphosphatase) ist ein ATP-powered Ion Pumpe in allen tierischen Zellen gefunden. Es ist verantwortlich für die Aufrechterhaltung der hohen Konzentration von Kalium-Ionen und niedrige Konzentration o Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-natrium-kalium-ionen-pumpe-protein-molekulmodell-natrium-kalium-atpase-adenosin-triphosphatase-ist-ein-atp-powered-ion-pumpe-in-allen-tierischen-zellen-gefunden-es-ist-verantwortlich-fur-die-aufrechterhaltung-der-hohen-konzentration-von-kalium-ionen-und-niedrige-konzentration-o-73688275.htmlRFE7TP43–Natrium-Kalium-Ionen Pumpe Protein, Molekülmodell. Natrium-Kalium-ATPase (Adenosin Triphosphatase) ist ein ATP-powered Ion Pumpe in allen tierischen Zellen gefunden. Es ist verantwortlich für die Aufrechterhaltung der hohen Konzentration von Kalium-Ionen und niedrige Konzentration o
 Laktat-Dehydrogenase Enzym, Molekülmodell. Dieses Enzym wandelt Pyruvat zu Laktat in die letzten Schritte der Glykolyse, Laktat, Pyruvat während des Milchsäure-Zyklus. Glykolyse ist der Stoffwechselweg, der Energie aus Glukose produziert. Die lact Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-laktat-dehydrogenase-enzym-molekulmodell-dieses-enzym-wandelt-pyruvat-zu-laktat-in-die-letzten-schritte-der-glykolyse-laktat-pyruvat-wahrend-des-milchsaure-zyklus-glykolyse-ist-der-stoffwechselweg-der-energie-aus-glukose-produziert-die-lact-73688318.html
Laktat-Dehydrogenase Enzym, Molekülmodell. Dieses Enzym wandelt Pyruvat zu Laktat in die letzten Schritte der Glykolyse, Laktat, Pyruvat während des Milchsäure-Zyklus. Glykolyse ist der Stoffwechselweg, der Energie aus Glukose produziert. Die lact Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-laktat-dehydrogenase-enzym-molekulmodell-dieses-enzym-wandelt-pyruvat-zu-laktat-in-die-letzten-schritte-der-glykolyse-laktat-pyruvat-wahrend-des-milchsaure-zyklus-glykolyse-ist-der-stoffwechselweg-der-energie-aus-glukose-produziert-die-lact-73688318.htmlRFE7TP5J–Laktat-Dehydrogenase Enzym, Molekülmodell. Dieses Enzym wandelt Pyruvat zu Laktat in die letzten Schritte der Glykolyse, Laktat, Pyruvat während des Milchsäure-Zyklus. Glykolyse ist der Stoffwechselweg, der Energie aus Glukose produziert. Die lact
 Integrin transmembrane Domäne, Molekülmodell. Integrine sind transmembranen Zellrezeptoren Adhäsion. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-integrin-transmembrane-domane-molekulmodell-integrine-sind-transmembranen-zellrezeptoren-adhasion-73688128.html
Integrin transmembrane Domäne, Molekülmodell. Integrine sind transmembranen Zellrezeptoren Adhäsion. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-integrin-transmembrane-domane-molekulmodell-integrine-sind-transmembranen-zellrezeptoren-adhasion-73688128.htmlRFE7TNXT–Integrin transmembrane Domäne, Molekülmodell. Integrine sind transmembranen Zellrezeptoren Adhäsion.
 Programmierten Zelle Tod Protein 6, Molekülmodell. Dies ist ein Kalzium-bindende Protein engagiert sich bei der Apoptose (programmierter Zelltod). Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-programmierten-zelle-tod-protein-6-molekulmodell-dies-ist-ein-kalzium-bindende-protein-engagiert-sich-bei-der-apoptose-programmierter-zelltod-73687959.html
Programmierten Zelle Tod Protein 6, Molekülmodell. Dies ist ein Kalzium-bindende Protein engagiert sich bei der Apoptose (programmierter Zelltod). Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-programmierten-zelle-tod-protein-6-molekulmodell-dies-ist-ein-kalzium-bindende-protein-engagiert-sich-bei-der-apoptose-programmierter-zelltod-73687959.htmlRFE7TNMR–Programmierten Zelle Tod Protein 6, Molekülmodell. Dies ist ein Kalzium-bindende Protein engagiert sich bei der Apoptose (programmierter Zelltod).
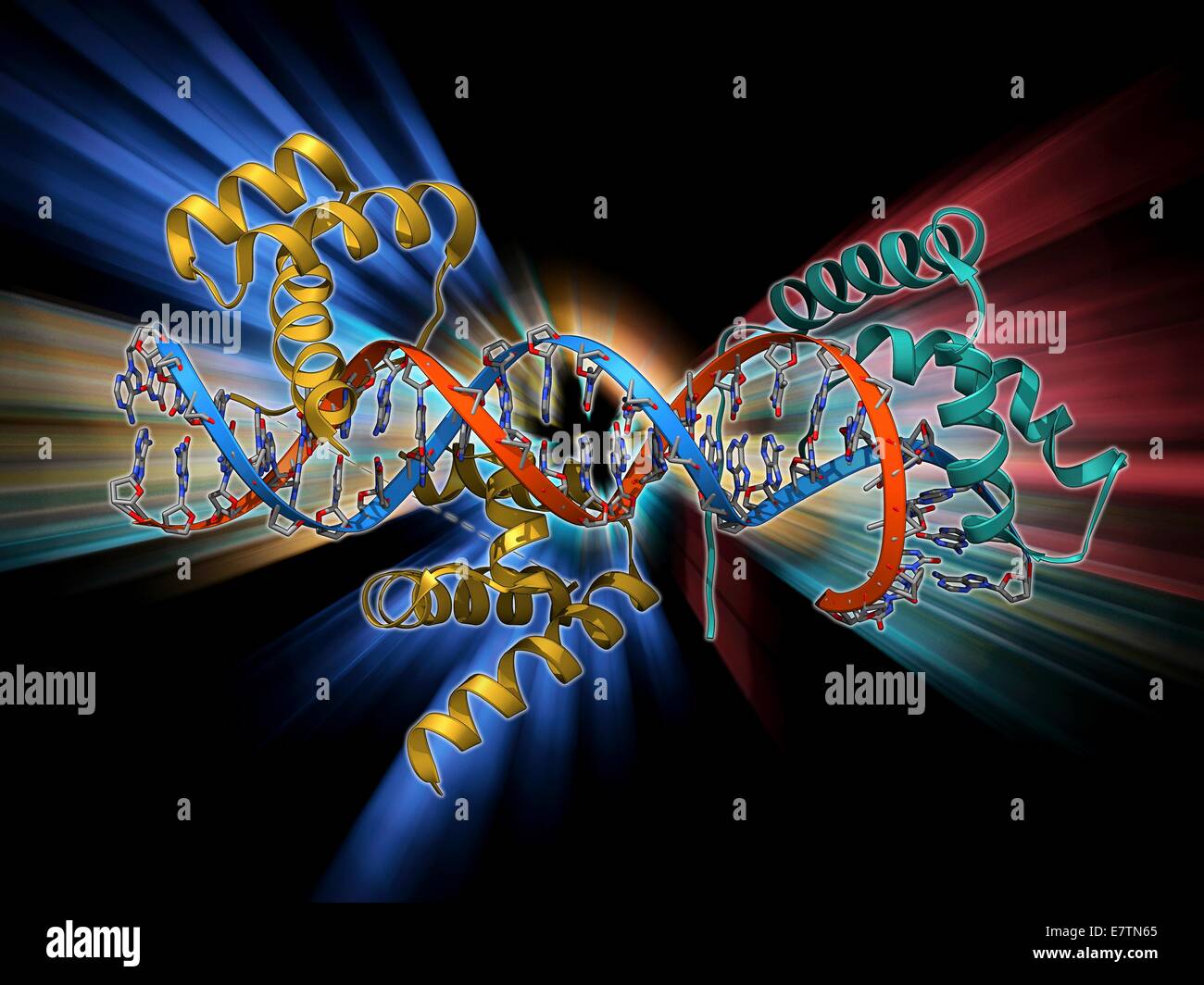 Transkriptionsfaktoren an DNA gebunden. Molekülmodell der Oct4 (rosa) und (grün) Transkriptionsfaktoren Sox2 gebunden an ein Molekül der DNA (Desoxyribonukleinsäure, rot und blau). Transkriptionsfaktoren sind Proteine, die an spezifische Sequenzen von DNA und c binden Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-transkriptionsfaktoren-an-dna-gebunden-molekulmodell-der-oct4-rosa-und-grun-transkriptionsfaktoren-sox2-gebunden-an-ein-molekul-der-dna-desoxyribonukleinsaure-rot-und-blau-transkriptionsfaktoren-sind-proteine-die-an-spezifische-sequenzen-von-dna-und-c-binden-73687549.html
Transkriptionsfaktoren an DNA gebunden. Molekülmodell der Oct4 (rosa) und (grün) Transkriptionsfaktoren Sox2 gebunden an ein Molekül der DNA (Desoxyribonukleinsäure, rot und blau). Transkriptionsfaktoren sind Proteine, die an spezifische Sequenzen von DNA und c binden Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-transkriptionsfaktoren-an-dna-gebunden-molekulmodell-der-oct4-rosa-und-grun-transkriptionsfaktoren-sox2-gebunden-an-ein-molekul-der-dna-desoxyribonukleinsaure-rot-und-blau-transkriptionsfaktoren-sind-proteine-die-an-spezifische-sequenzen-von-dna-und-c-binden-73687549.htmlRFE7TN65–Transkriptionsfaktoren an DNA gebunden. Molekülmodell der Oct4 (rosa) und (grün) Transkriptionsfaktoren Sox2 gebunden an ein Molekül der DNA (Desoxyribonukleinsäure, rot und blau). Transkriptionsfaktoren sind Proteine, die an spezifische Sequenzen von DNA und c binden
 Stickoxid-Synthase, Molekülmodell. Dieses Enzym katalysiert die Produktion von Stickoxid aus L-Arginin. Stickstoffmonoxid ist an zellulären Signalgebung beteiligt. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-stickoxid-synthase-molekulmodell-dieses-enzym-katalysiert-die-produktion-von-stickoxid-aus-l-arginin-stickstoffmonoxid-ist-an-zellularen-signalgebung-beteiligt-73687641.html
Stickoxid-Synthase, Molekülmodell. Dieses Enzym katalysiert die Produktion von Stickoxid aus L-Arginin. Stickstoffmonoxid ist an zellulären Signalgebung beteiligt. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-stickoxid-synthase-molekulmodell-dieses-enzym-katalysiert-die-produktion-von-stickoxid-aus-l-arginin-stickstoffmonoxid-ist-an-zellularen-signalgebung-beteiligt-73687641.htmlRFE7TN9D–Stickoxid-Synthase, Molekülmodell. Dieses Enzym katalysiert die Produktion von Stickoxid aus L-Arginin. Stickstoffmonoxid ist an zellulären Signalgebung beteiligt.
 Uroporphyrinogen III Decarboxylase. Molekulares Modell des Enzyms menschlichen Uroporphyrinogen III Decarboxylase (UROD). Mutationen oder Mängel an diesem Enzym dazu führen, dass die Porphyrie Störung mit einem Build bis der Porphyrine im Körper. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-uroporphyrinogen-iii-decarboxylase-molekulares-modell-des-enzyms-menschlichen-uroporphyrinogen-iii-decarboxylase-urod-mutationen-oder-mangel-an-diesem-enzym-dazu-fuhren-dass-die-porphyrie-storung-mit-einem-build-bis-der-porphyrine-im-korper-73687589.html
Uroporphyrinogen III Decarboxylase. Molekulares Modell des Enzyms menschlichen Uroporphyrinogen III Decarboxylase (UROD). Mutationen oder Mängel an diesem Enzym dazu führen, dass die Porphyrie Störung mit einem Build bis der Porphyrine im Körper. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-uroporphyrinogen-iii-decarboxylase-molekulares-modell-des-enzyms-menschlichen-uroporphyrinogen-iii-decarboxylase-urod-mutationen-oder-mangel-an-diesem-enzym-dazu-fuhren-dass-die-porphyrie-storung-mit-einem-build-bis-der-porphyrine-im-korper-73687589.htmlRFE7TN7H–Uroporphyrinogen III Decarboxylase. Molekulares Modell des Enzyms menschlichen Uroporphyrinogen III Decarboxylase (UROD). Mutationen oder Mängel an diesem Enzym dazu führen, dass die Porphyrie Störung mit einem Build bis der Porphyrine im Körper.
 Menschlichen Vorläufer Prionprotein, Molekülmodell zeigt Sekundärstruktur. Die menschlichen Prion-Protein (hPrP) ist ein Prion-Vorläufer. Prionen sind abnorme Proteine, die die Ursache der transmissiblen spongiformen Enzephalopathien (TSE) sind wie BSE bei Kühen, Schrott Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-menschlichen-vorlaufer-prionprotein-molekulmodell-zeigt-sekundarstruktur-die-menschlichen-prion-protein-hprp-ist-ein-prion-vorlaufer-prionen-sind-abnorme-proteine-die-die-ursache-der-transmissiblen-spongiformen-enzephalopathien-tse-sind-wie-bse-bei-kuhen-schrott-73687557.html
Menschlichen Vorläufer Prionprotein, Molekülmodell zeigt Sekundärstruktur. Die menschlichen Prion-Protein (hPrP) ist ein Prion-Vorläufer. Prionen sind abnorme Proteine, die die Ursache der transmissiblen spongiformen Enzephalopathien (TSE) sind wie BSE bei Kühen, Schrott Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-menschlichen-vorlaufer-prionprotein-molekulmodell-zeigt-sekundarstruktur-die-menschlichen-prion-protein-hprp-ist-ein-prion-vorlaufer-prionen-sind-abnorme-proteine-die-die-ursache-der-transmissiblen-spongiformen-enzephalopathien-tse-sind-wie-bse-bei-kuhen-schrott-73687557.htmlRFE7TN6D–Menschlichen Vorläufer Prionprotein, Molekülmodell zeigt Sekundärstruktur. Die menschlichen Prion-Protein (hPrP) ist ein Prion-Vorläufer. Prionen sind abnorme Proteine, die die Ursache der transmissiblen spongiformen Enzephalopathien (TSE) sind wie BSE bei Kühen, Schrott
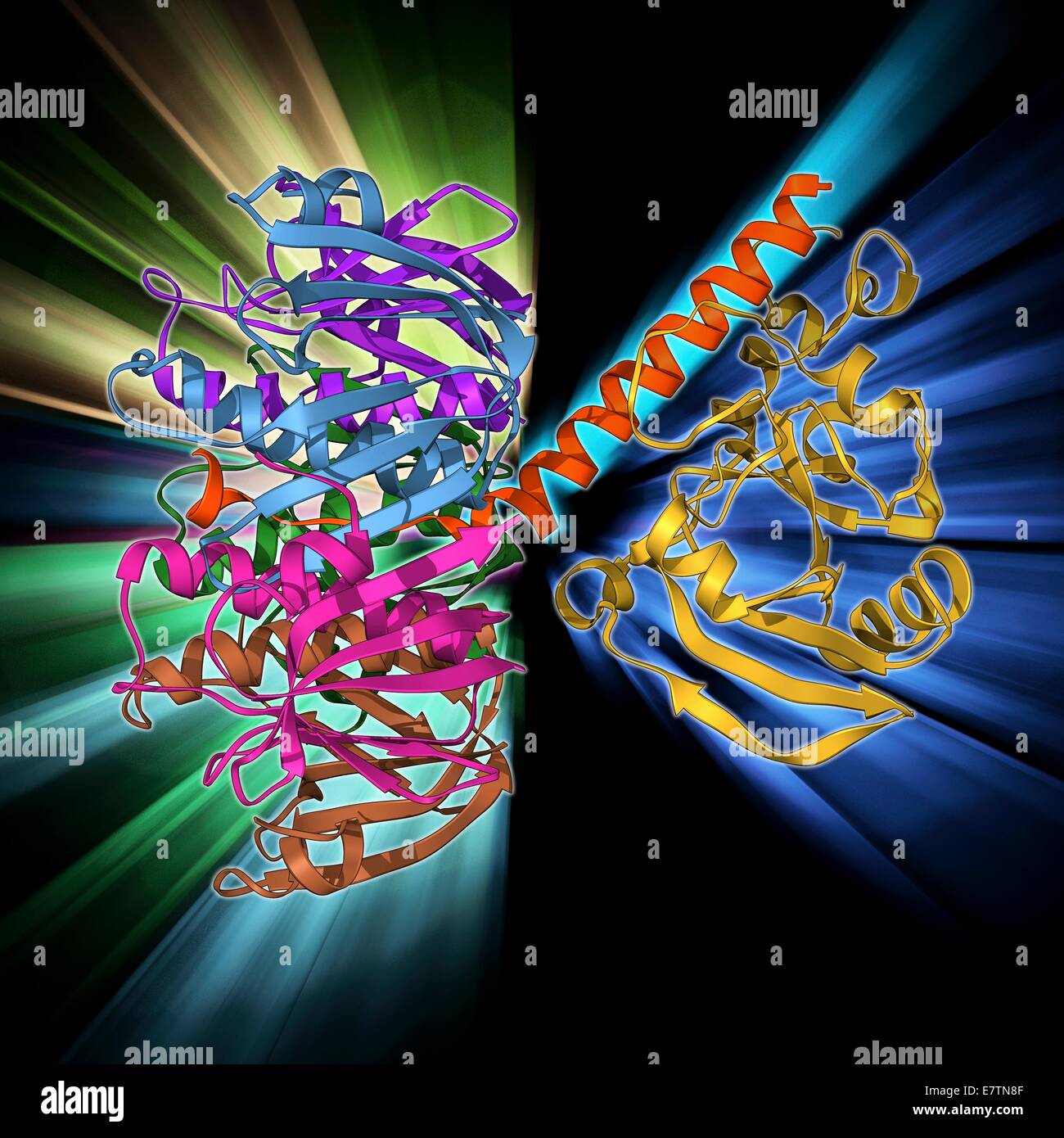 Escherichia coli Hitze-labile Enterotoxin, Molekülmodell. Dies ist einer von mehreren Proteinen von pathogenen E. Coli-Bakterien im Darm produziert. Im Gegensatz zu den hitzebeständig Enterotoxin wird dieser an den Hochtemperaturen inaktiviert. Das Toxin bewirkt, dass diarrh Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-escherichia-coli-hitze-labile-enterotoxin-molekulmodell-dies-ist-einer-von-mehreren-proteinen-von-pathogenen-e-coli-bakterien-im-darm-produziert-im-gegensatz-zu-den-hitzebestandig-enterotoxin-wird-dieser-an-den-hochtemperaturen-inaktiviert-das-toxin-bewirkt-dass-diarrh-73687615.html
Escherichia coli Hitze-labile Enterotoxin, Molekülmodell. Dies ist einer von mehreren Proteinen von pathogenen E. Coli-Bakterien im Darm produziert. Im Gegensatz zu den hitzebeständig Enterotoxin wird dieser an den Hochtemperaturen inaktiviert. Das Toxin bewirkt, dass diarrh Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-escherichia-coli-hitze-labile-enterotoxin-molekulmodell-dies-ist-einer-von-mehreren-proteinen-von-pathogenen-e-coli-bakterien-im-darm-produziert-im-gegensatz-zu-den-hitzebestandig-enterotoxin-wird-dieser-an-den-hochtemperaturen-inaktiviert-das-toxin-bewirkt-dass-diarrh-73687615.htmlRFE7TN8F–Escherichia coli Hitze-labile Enterotoxin, Molekülmodell. Dies ist einer von mehreren Proteinen von pathogenen E. Coli-Bakterien im Darm produziert. Im Gegensatz zu den hitzebeständig Enterotoxin wird dieser an den Hochtemperaturen inaktiviert. Das Toxin bewirkt, dass diarrh
 Epidermalen Wachstumsfaktor-Molekül. Molekulares Modell des epidermalen Wachstumsfaktor (EGF) an einen Rezeptor gebunden. EGF spielt eine wichtige Rolle bei der Regulation des Zellwachstums, Proliferation und Differenzierung. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-epidermalen-wachstumsfaktor-molekul-molekulares-modell-des-epidermalen-wachstumsfaktor-egf-an-einen-rezeptor-gebunden-egf-spielt-eine-wichtige-rolle-bei-der-regulation-des-zellwachstums-proliferation-und-differenzierung-73687645.html
Epidermalen Wachstumsfaktor-Molekül. Molekulares Modell des epidermalen Wachstumsfaktor (EGF) an einen Rezeptor gebunden. EGF spielt eine wichtige Rolle bei der Regulation des Zellwachstums, Proliferation und Differenzierung. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-epidermalen-wachstumsfaktor-molekul-molekulares-modell-des-epidermalen-wachstumsfaktor-egf-an-einen-rezeptor-gebunden-egf-spielt-eine-wichtige-rolle-bei-der-regulation-des-zellwachstums-proliferation-und-differenzierung-73687645.htmlRFE7TN9H–Epidermalen Wachstumsfaktor-Molekül. Molekulares Modell des epidermalen Wachstumsfaktor (EGF) an einen Rezeptor gebunden. EGF spielt eine wichtige Rolle bei der Regulation des Zellwachstums, Proliferation und Differenzierung.
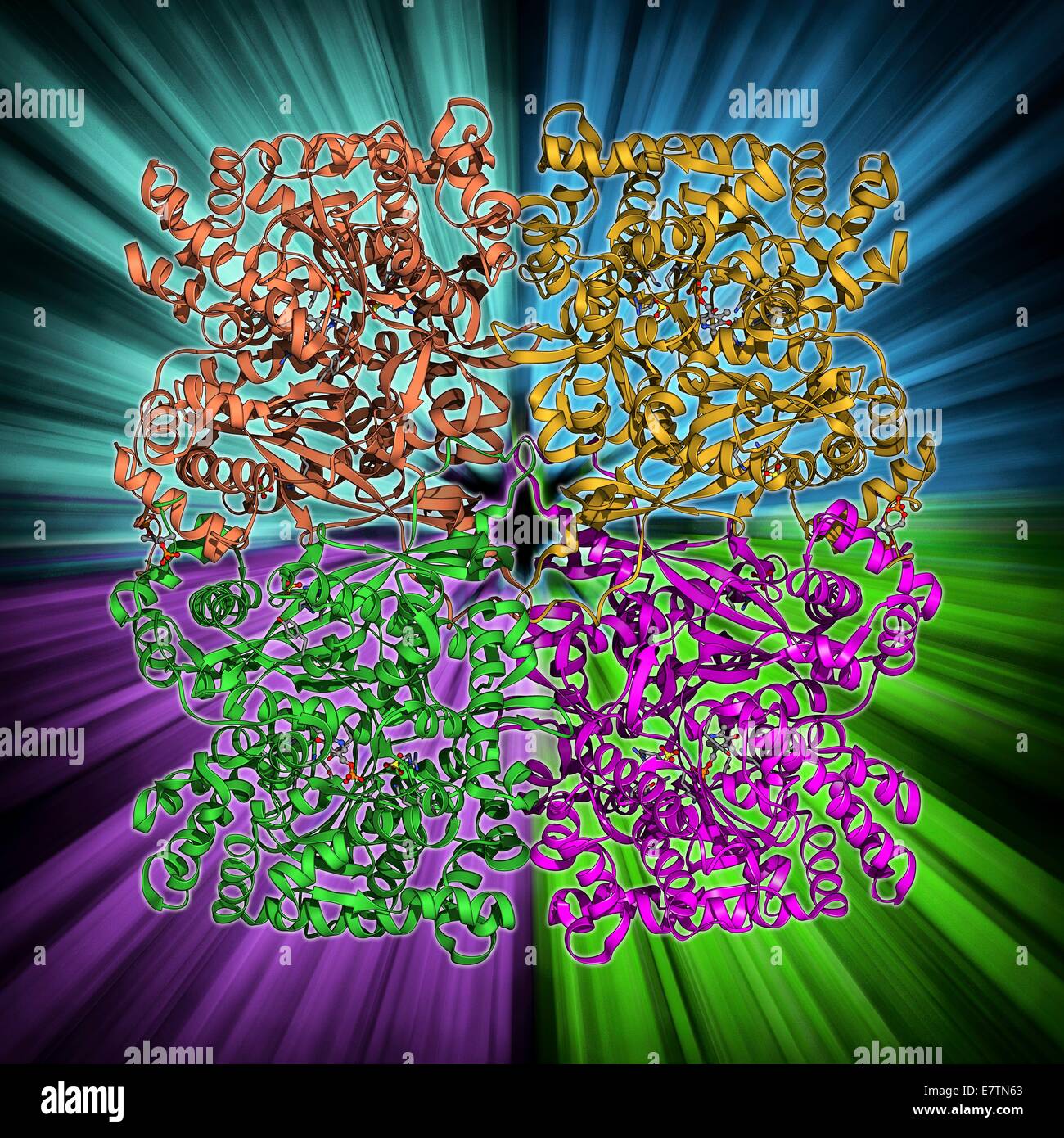 Glykogen-Phosphorylase, Molekülmodell. Dies ist ein Enzym, das beim Abbau von Glykogen, Lagerung Energiemolekül tierischen Stoffwechsel beteiligt. Die Wirkung der Glykogen-Phosphorylase liefert Kraftstoff für die Muskelkontraktion. In der Leber das Enzym m Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-glykogen-phosphorylase-molekulmodell-dies-ist-ein-enzym-das-beim-abbau-von-glykogen-lagerung-energiemolekul-tierischen-stoffwechsel-beteiligt-die-wirkung-der-glykogen-phosphorylase-liefert-kraftstoff-fur-die-muskelkontraktion-in-der-leber-das-enzym-m-73687547.html
Glykogen-Phosphorylase, Molekülmodell. Dies ist ein Enzym, das beim Abbau von Glykogen, Lagerung Energiemolekül tierischen Stoffwechsel beteiligt. Die Wirkung der Glykogen-Phosphorylase liefert Kraftstoff für die Muskelkontraktion. In der Leber das Enzym m Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-glykogen-phosphorylase-molekulmodell-dies-ist-ein-enzym-das-beim-abbau-von-glykogen-lagerung-energiemolekul-tierischen-stoffwechsel-beteiligt-die-wirkung-der-glykogen-phosphorylase-liefert-kraftstoff-fur-die-muskelkontraktion-in-der-leber-das-enzym-m-73687547.htmlRFE7TN63–Glykogen-Phosphorylase, Molekülmodell. Dies ist ein Enzym, das beim Abbau von Glykogen, Lagerung Energiemolekül tierischen Stoffwechsel beteiligt. Die Wirkung der Glykogen-Phosphorylase liefert Kraftstoff für die Muskelkontraktion. In der Leber das Enzym m
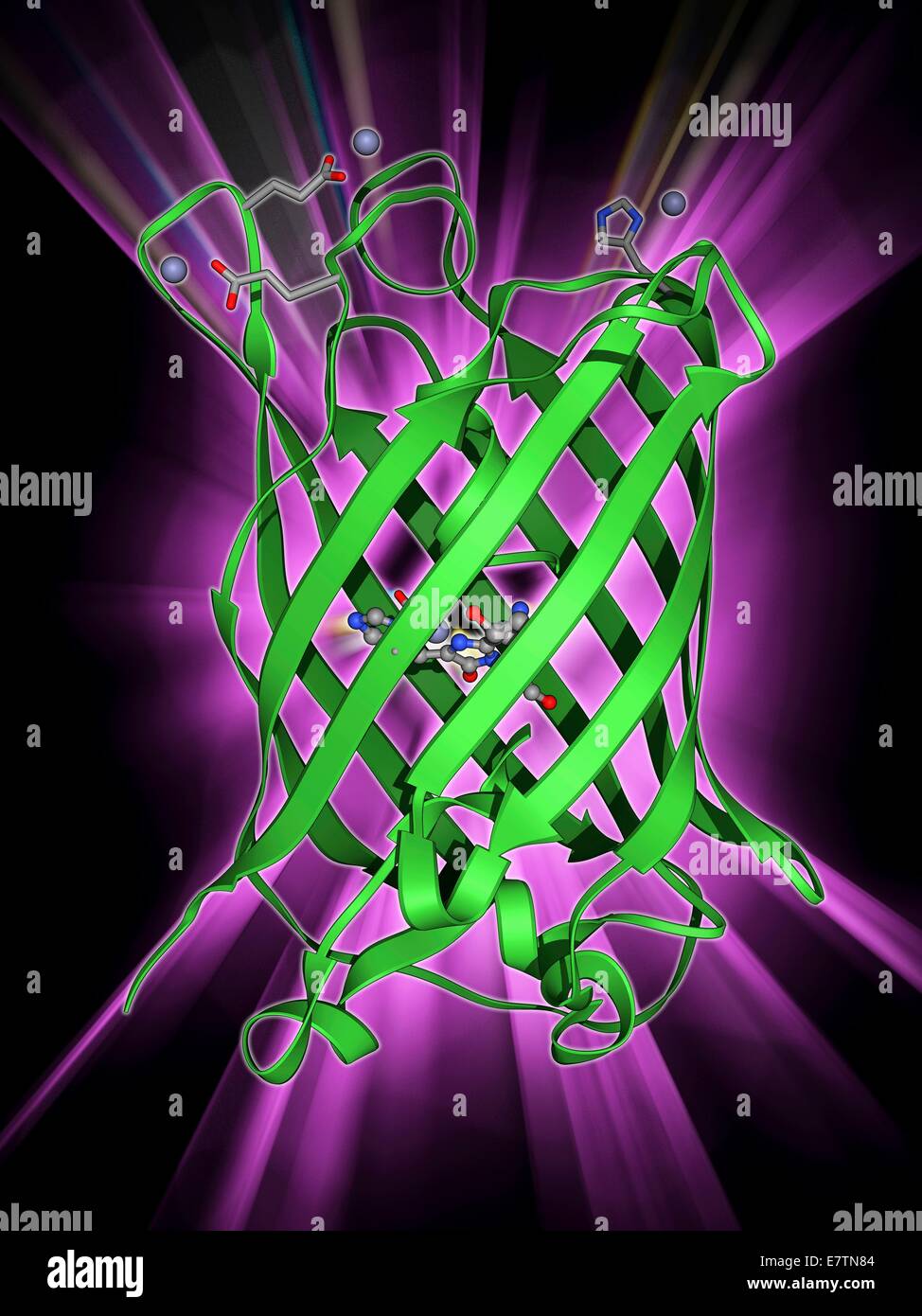 Grün fluoreszierendes Protein (GFP), molekulare Modell. Das Molekül hat eine zylindrische Struktur, die aus Beta-Scheiben (Bänder) gebildet. GFP ist in das Pazifische Qualle Aequorea Victoria gefunden. Es fluoresziert grün als blaues Licht darauf schien ist. GFP ist am meisten benutzt ein Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-grun-fluoreszierendes-protein-gfp-molekulare-modell-das-molekul-hat-eine-zylindrische-struktur-die-aus-beta-scheiben-bander-gebildet-gfp-ist-in-das-pazifische-qualle-aequorea-victoria-gefunden-es-fluoresziert-grun-als-blaues-licht-darauf-schien-ist-gfp-ist-am-meisten-benutzt-ein-73687604.html
Grün fluoreszierendes Protein (GFP), molekulare Modell. Das Molekül hat eine zylindrische Struktur, die aus Beta-Scheiben (Bänder) gebildet. GFP ist in das Pazifische Qualle Aequorea Victoria gefunden. Es fluoresziert grün als blaues Licht darauf schien ist. GFP ist am meisten benutzt ein Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-grun-fluoreszierendes-protein-gfp-molekulare-modell-das-molekul-hat-eine-zylindrische-struktur-die-aus-beta-scheiben-bander-gebildet-gfp-ist-in-das-pazifische-qualle-aequorea-victoria-gefunden-es-fluoresziert-grun-als-blaues-licht-darauf-schien-ist-gfp-ist-am-meisten-benutzt-ein-73687604.htmlRFE7TN84–Grün fluoreszierendes Protein (GFP), molekulare Modell. Das Molekül hat eine zylindrische Struktur, die aus Beta-Scheiben (Bänder) gebildet. GFP ist in das Pazifische Qualle Aequorea Victoria gefunden. Es fluoresziert grün als blaues Licht darauf schien ist. GFP ist am meisten benutzt ein
 Dehnung Faktor G. Molecular Modell der Dehnung Faktor G (EF-G) komplexiert mit BIP (Guanosin diphosphat). Dieses Enzym ist an die Dehnung der Polypeptidketten während der Übersetzung, die Produktion eines Proteins aus einer mRNA (Messenger Ribonucl beteiligt Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-dehnung-faktor-g-molecular-modell-der-dehnung-faktor-g-ef-g-komplexiert-mit-bip-guanosin-diphosphat-dieses-enzym-ist-an-die-dehnung-der-polypeptidketten-wahrend-der-ubersetzung-die-produktion-eines-proteins-aus-einer-mrna-messenger-ribonucl-beteiligt-73687479.html
Dehnung Faktor G. Molecular Modell der Dehnung Faktor G (EF-G) komplexiert mit BIP (Guanosin diphosphat). Dieses Enzym ist an die Dehnung der Polypeptidketten während der Übersetzung, die Produktion eines Proteins aus einer mRNA (Messenger Ribonucl beteiligt Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-dehnung-faktor-g-molecular-modell-der-dehnung-faktor-g-ef-g-komplexiert-mit-bip-guanosin-diphosphat-dieses-enzym-ist-an-die-dehnung-der-polypeptidketten-wahrend-der-ubersetzung-die-produktion-eines-proteins-aus-einer-mrna-messenger-ribonucl-beteiligt-73687479.htmlRFE7TN3K–Dehnung Faktor G. Molecular Modell der Dehnung Faktor G (EF-G) komplexiert mit BIP (Guanosin diphosphat). Dieses Enzym ist an die Dehnung der Polypeptidketten während der Übersetzung, die Produktion eines Proteins aus einer mRNA (Messenger Ribonucl beteiligt
 Valyl-tRNA Synthestase Eiweißmolekül. Molekülmodell zeigt bakterielle Valyl-tRNA Synthestase komplexiert mit Valyl tRNA (Transfer-Ribonukleinsäure). Valyl-tRNA Synthestase legt die richtige Aminosäure, in diesem Fall Valyl, auf die entsprechende tRNA m Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-valyl-trna-synthestase-eiweissmolekul-molekulmodell-zeigt-bakterielle-valyl-trna-synthestase-komplexiert-mit-valyl-trna-transfer-ribonukleinsaure-valyl-trna-synthestase-legt-die-richtige-aminosaure-in-diesem-fall-valyl-auf-die-entsprechende-trna-m-73687542.html
Valyl-tRNA Synthestase Eiweißmolekül. Molekülmodell zeigt bakterielle Valyl-tRNA Synthestase komplexiert mit Valyl tRNA (Transfer-Ribonukleinsäure). Valyl-tRNA Synthestase legt die richtige Aminosäure, in diesem Fall Valyl, auf die entsprechende tRNA m Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-valyl-trna-synthestase-eiweissmolekul-molekulmodell-zeigt-bakterielle-valyl-trna-synthestase-komplexiert-mit-valyl-trna-transfer-ribonukleinsaure-valyl-trna-synthestase-legt-die-richtige-aminosaure-in-diesem-fall-valyl-auf-die-entsprechende-trna-m-73687542.htmlRFE7TN5X–Valyl-tRNA Synthestase Eiweißmolekül. Molekülmodell zeigt bakterielle Valyl-tRNA Synthestase komplexiert mit Valyl tRNA (Transfer-Ribonukleinsäure). Valyl-tRNA Synthestase legt die richtige Aminosäure, in diesem Fall Valyl, auf die entsprechende tRNA m
 Semliki Forest Virus Kapsid, Molekülmodell. Dieses Virus, benannt nach dem Wald in Uganda, wo es identifiziert wurde, ist durch den Stich von Mücken verbreitet. Es kann sowohl Menschen als auch Tiere infizieren. Viren wird das Kapsid die Protein-Shell, die das Gen umschließt Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-semliki-forest-virus-kapsid-molekulmodell-dieses-virus-benannt-nach-dem-wald-in-uganda-wo-es-identifiziert-wurde-ist-durch-den-stich-von-mucken-verbreitet-es-kann-sowohl-menschen-als-auch-tiere-infizieren-viren-wird-das-kapsid-die-protein-shell-die-das-gen-umschliesst-73687493.html
Semliki Forest Virus Kapsid, Molekülmodell. Dieses Virus, benannt nach dem Wald in Uganda, wo es identifiziert wurde, ist durch den Stich von Mücken verbreitet. Es kann sowohl Menschen als auch Tiere infizieren. Viren wird das Kapsid die Protein-Shell, die das Gen umschließt Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-semliki-forest-virus-kapsid-molekulmodell-dieses-virus-benannt-nach-dem-wald-in-uganda-wo-es-identifiziert-wurde-ist-durch-den-stich-von-mucken-verbreitet-es-kann-sowohl-menschen-als-auch-tiere-infizieren-viren-wird-das-kapsid-die-protein-shell-die-das-gen-umschliesst-73687493.htmlRFE7TN45–Semliki Forest Virus Kapsid, Molekülmodell. Dieses Virus, benannt nach dem Wald in Uganda, wo es identifiziert wurde, ist durch den Stich von Mücken verbreitet. Es kann sowohl Menschen als auch Tiere infizieren. Viren wird das Kapsid die Protein-Shell, die das Gen umschließt
 Synthetische dreifache schraubenartige Peptid, Molekülmodell. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-synthetische-dreifache-schraubenartige-peptid-molekulmodell-73687469.html
Synthetische dreifache schraubenartige Peptid, Molekülmodell. Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-synthetische-dreifache-schraubenartige-peptid-molekulmodell-73687469.htmlRFE7TN39–Synthetische dreifache schraubenartige Peptid, Molekülmodell.
 Cyclohexanon Molekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-cyclohexanon-molekul-47691555.html
Cyclohexanon Molekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-cyclohexanon-molekul-47691555.htmlRFCNGF1R–Cyclohexanon Molekül
 Atazanavir Wirkstoffmolekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-atazanavir-wirkstoffmolekul-47691159.html
Atazanavir Wirkstoffmolekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-atazanavir-wirkstoffmolekul-47691159.htmlRFCNGEFK–Atazanavir Wirkstoffmolekül
 Cisplatin Wirkstoffmolekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-cisplatin-wirkstoffmolekul-47691466.html
Cisplatin Wirkstoffmolekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-cisplatin-wirkstoffmolekul-47691466.htmlRFCNGEXJ–Cisplatin Wirkstoffmolekül
 Letrozol Wirkstoffmolekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-letrozol-wirkstoffmolekul-47691947.html
Letrozol Wirkstoffmolekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-letrozol-wirkstoffmolekul-47691947.htmlRFCNGFFR–Letrozol Wirkstoffmolekül
 Capecitabin Wirkstoffmolekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-capecitabin-wirkstoffmolekul-47691288.html
Capecitabin Wirkstoffmolekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-capecitabin-wirkstoffmolekul-47691288.htmlRFCNGEM8–Capecitabin Wirkstoffmolekül
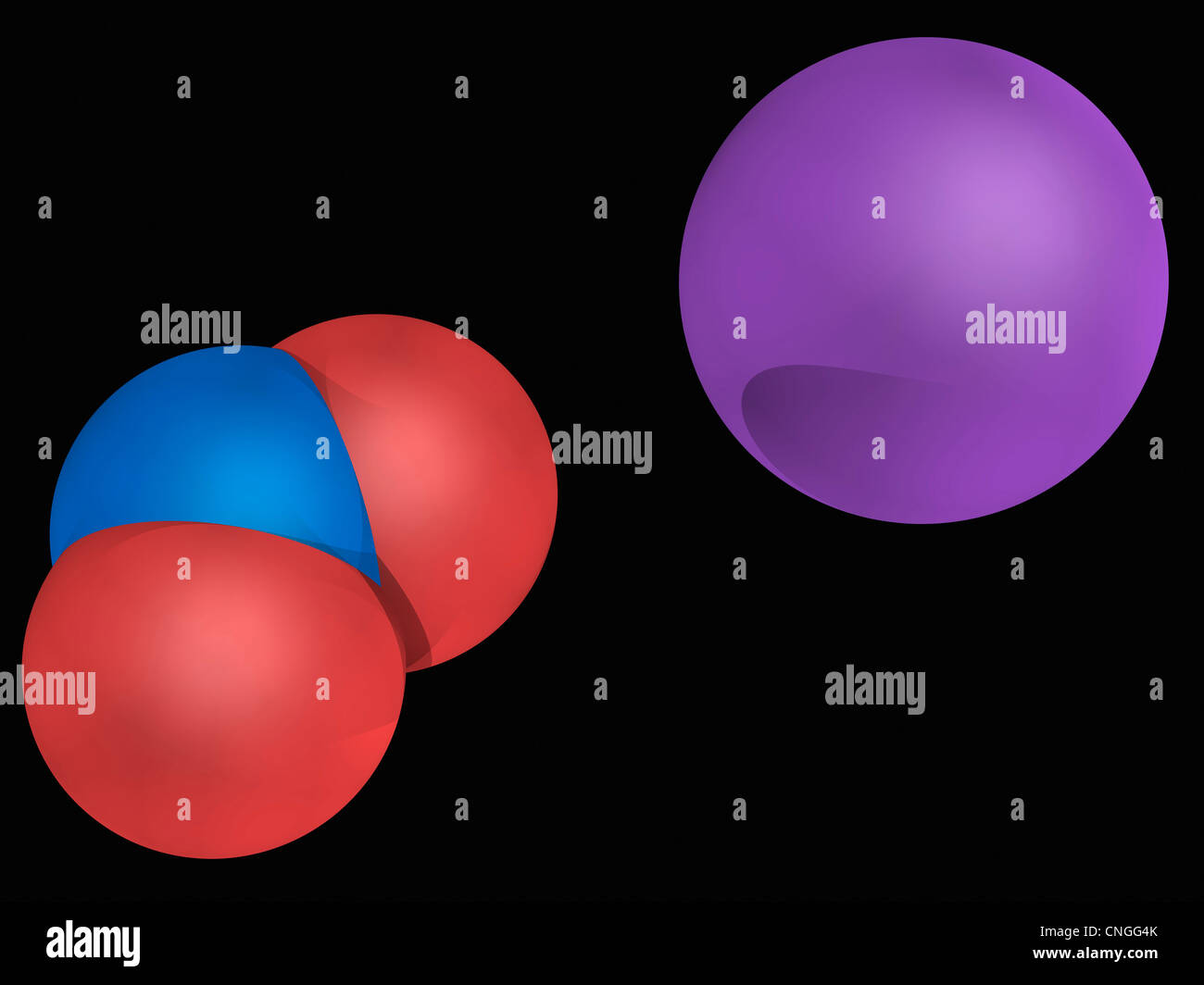 Natrium-Nitrit-Molekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-natrium-nitrit-molekul-47692419.html
Natrium-Nitrit-Molekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-natrium-nitrit-molekul-47692419.htmlRFCNGG4K–Natrium-Nitrit-Molekül
 Metaxalone Wirkstoffmolekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-metaxalone-wirkstoffmolekul-47691984.html
Metaxalone Wirkstoffmolekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-metaxalone-wirkstoffmolekul-47691984.htmlRFCNGFH4–Metaxalone Wirkstoffmolekül
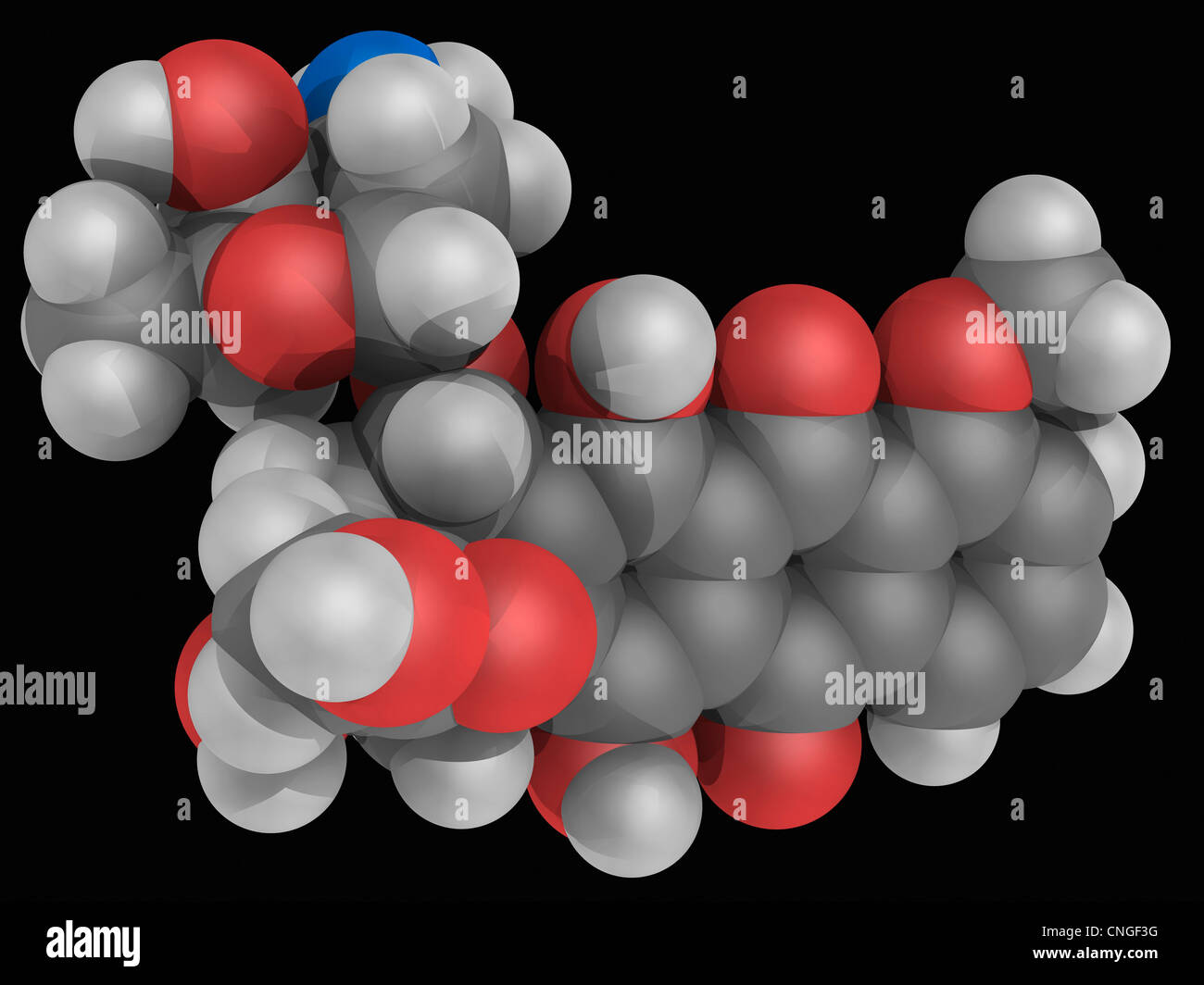 Doxorubicin Wirkstoffmolekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-doxorubicin-wirkstoffmolekul-47691604.html
Doxorubicin Wirkstoffmolekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-doxorubicin-wirkstoffmolekul-47691604.htmlRFCNGF3G–Doxorubicin Wirkstoffmolekül
 Amygdalin-Molekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-amygdalin-molekul-47691102.html
Amygdalin-Molekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-amygdalin-molekul-47691102.htmlRFCNGEDJ–Amygdalin-Molekül
 Lidocain Wirkstoffmolekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-lidocain-wirkstoffmolekul-47691979.html
Lidocain Wirkstoffmolekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-lidocain-wirkstoffmolekul-47691979.htmlRFCNGFGY–Lidocain Wirkstoffmolekül
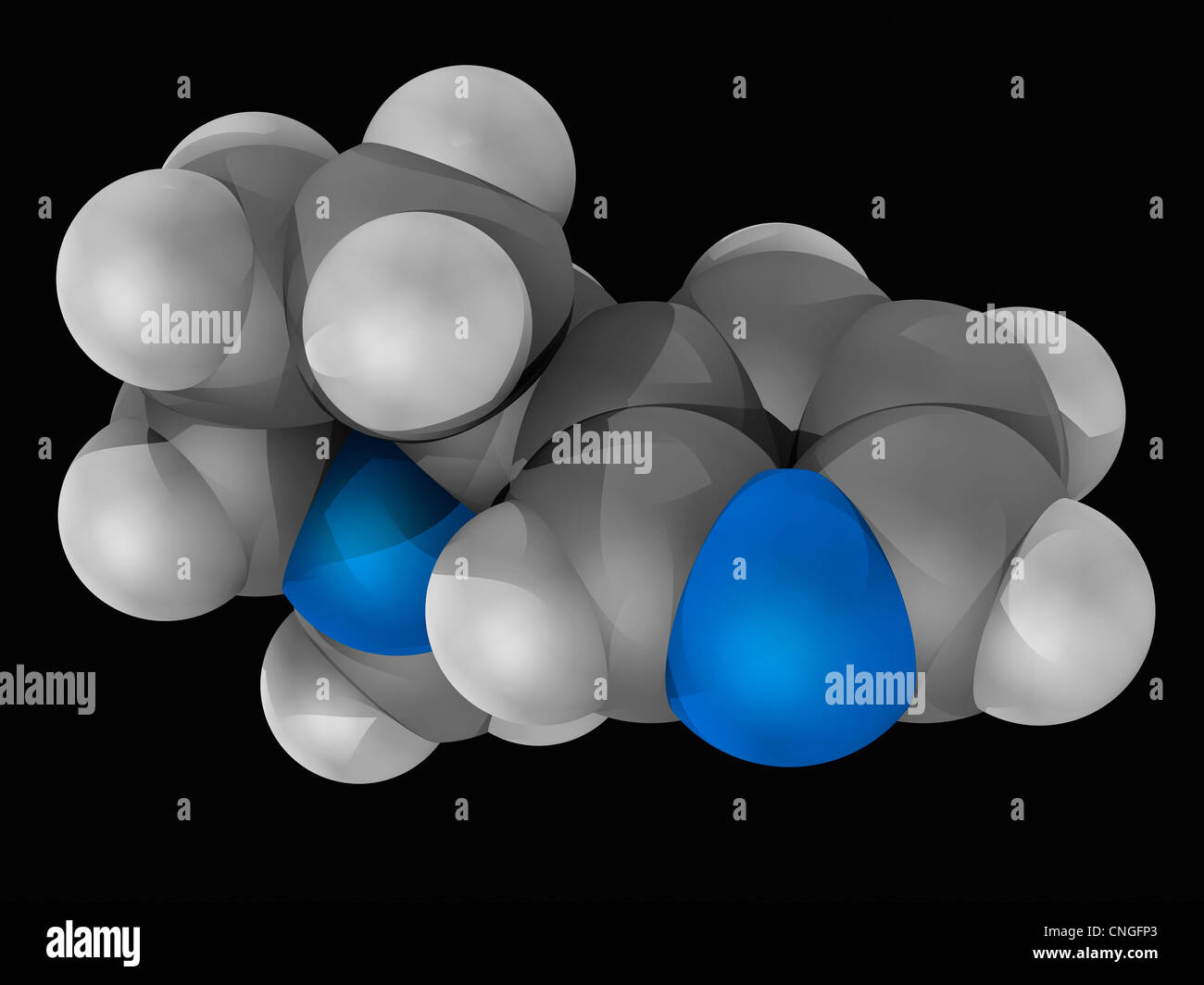 Nikotin-Molekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-nikotin-molekul-47692123.html
Nikotin-Molekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-nikotin-molekul-47692123.htmlRFCNGFP3–Nikotin-Molekül
 Methylprednisolon Wirkstoffmolekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-methylprednisolon-wirkstoffmolekul-47692021.html
Methylprednisolon Wirkstoffmolekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-methylprednisolon-wirkstoffmolekul-47692021.htmlRFCNGFJD–Methylprednisolon Wirkstoffmolekül
 Acetol Molekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-acetol-molekul-47691091.html
Acetol Molekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-acetol-molekul-47691091.htmlRFCNGED7–Acetol Molekül
 Paracetamol-Wirkstoffmolekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-paracetamol-wirkstoffmolekul-47692208.html
Paracetamol-Wirkstoffmolekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-paracetamol-wirkstoffmolekul-47692208.htmlRFCNGFW4–Paracetamol-Wirkstoffmolekül
 Cefuroxim Wirkstoffmolekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-cefuroxim-wirkstoffmolekul-47691366.html
Cefuroxim Wirkstoffmolekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-cefuroxim-wirkstoffmolekul-47691366.htmlRFCNGER2–Cefuroxim Wirkstoffmolekül
 Latanoprost Wirkstoffmolekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-latanoprost-wirkstoffmolekul-47691940.html
Latanoprost Wirkstoffmolekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-latanoprost-wirkstoffmolekul-47691940.htmlRFCNGFFG–Latanoprost Wirkstoffmolekül
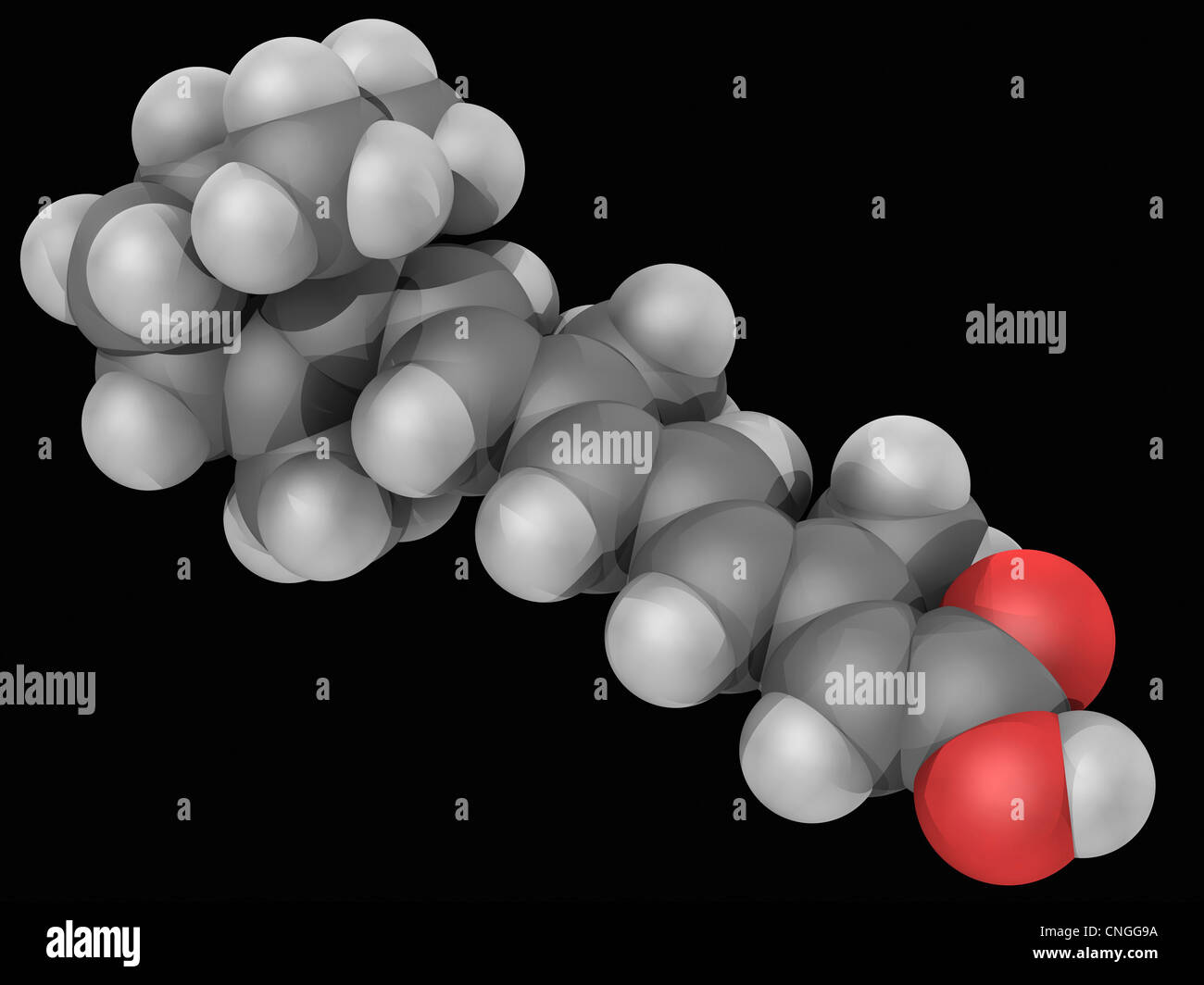 Tretinoin Wirkstoffmolekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-tretinoin-wirkstoffmolekul-47692550.html
Tretinoin Wirkstoffmolekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-tretinoin-wirkstoffmolekul-47692550.htmlRFCNGG9A–Tretinoin Wirkstoffmolekül
 Strychnin-Molekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-strychnin-molekul-47692443.html
Strychnin-Molekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-strychnin-molekul-47692443.htmlRFCNGG5F–Strychnin-Molekül
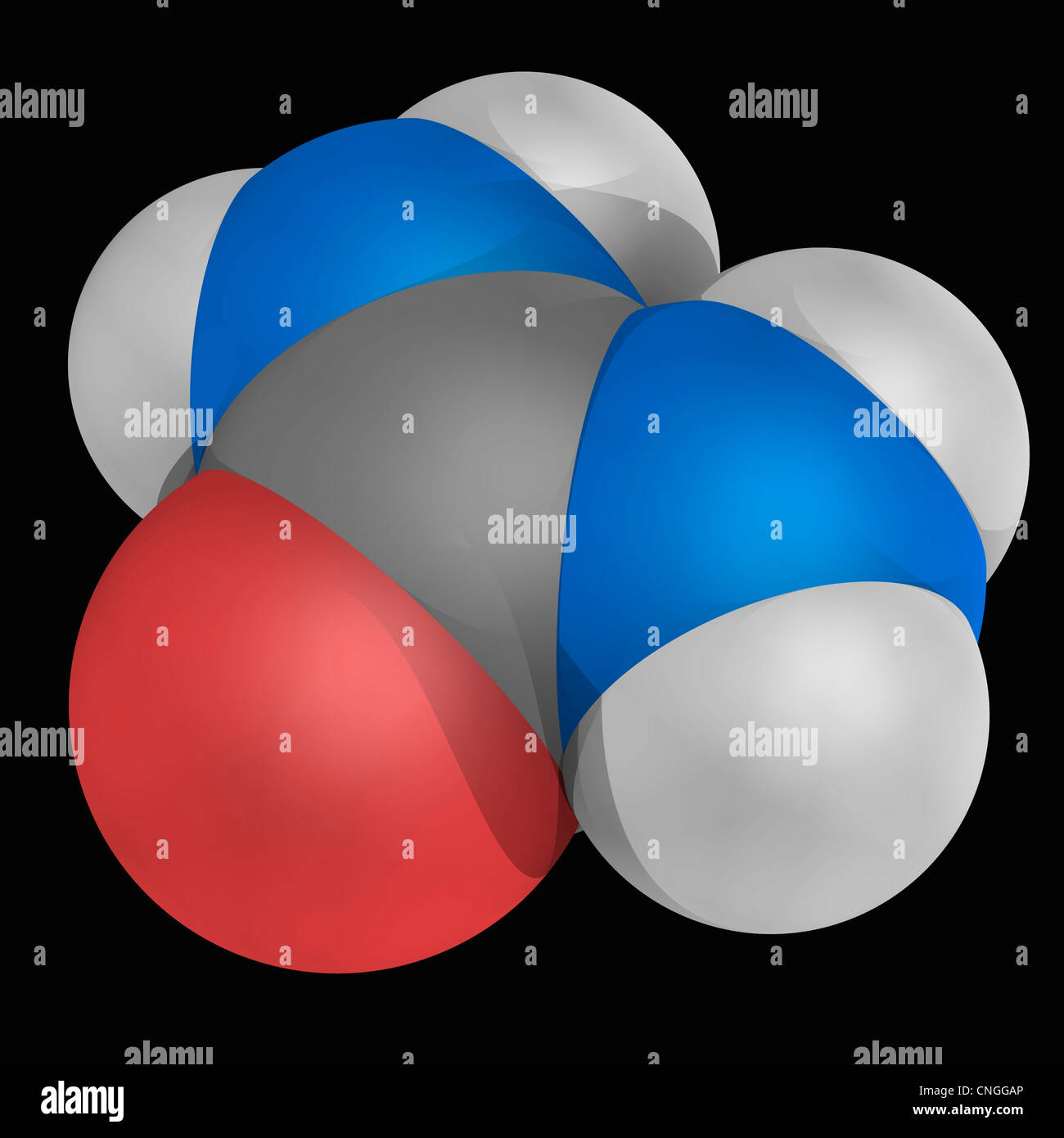 Harnstoff-Molekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-harnstoff-molekul-47692590.html
Harnstoff-Molekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-harnstoff-molekul-47692590.htmlRFCNGGAP–Harnstoff-Molekül
 Ondansetron Wirkstoffmolekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-ondansetron-wirkstoffmolekul-47692169.html
Ondansetron Wirkstoffmolekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-ondansetron-wirkstoffmolekul-47692169.htmlRFCNGFRN–Ondansetron Wirkstoffmolekül
 Uracil-Molekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-uracil-molekul-47692584.html
Uracil-Molekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-uracil-molekul-47692584.htmlRFCNGGAG–Uracil-Molekül
 Fosfomycin Wirkstoffmolekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-fosfomycin-wirkstoffmolekul-47691753.html
Fosfomycin Wirkstoffmolekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-fosfomycin-wirkstoffmolekul-47691753.htmlRFCNGF8W–Fosfomycin Wirkstoffmolekül
 Methoxyisoflavone Molekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-methoxyisoflavone-molekul-47692005.html
Methoxyisoflavone Molekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-methoxyisoflavone-molekul-47692005.htmlRFCNGFHW–Methoxyisoflavone Molekül
 Atorvastatin Wirkstoffmolekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-atorvastatin-wirkstoffmolekul-47691171.html
Atorvastatin Wirkstoffmolekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-atorvastatin-wirkstoffmolekul-47691171.htmlRFCNGEG3–Atorvastatin Wirkstoffmolekül
 Zolpidem Wirkstoffmolekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-zolpidem-wirkstoffmolekul-47692705.html
Zolpidem Wirkstoffmolekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-zolpidem-wirkstoffmolekul-47692705.htmlRFCNGGEW–Zolpidem Wirkstoffmolekül
 Brompheniramine Wirkstoffmolekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-brompheniramine-wirkstoffmolekul-47691262.html
Brompheniramine Wirkstoffmolekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-brompheniramine-wirkstoffmolekul-47691262.htmlRFCNGEKA–Brompheniramine Wirkstoffmolekül
 Penicillin G Wirkstoffmolekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-penicillin-g-wirkstoffmolekul-47692218.html
Penicillin G Wirkstoffmolekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-penicillin-g-wirkstoffmolekul-47692218.htmlRFCNGFWE–Penicillin G Wirkstoffmolekül
 Donepezil Wirkstoffmolekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-donepezil-wirkstoffmolekul-47691580.html
Donepezil Wirkstoffmolekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-donepezil-wirkstoffmolekul-47691580.htmlRFCNGF2M–Donepezil Wirkstoffmolekül
 Ecdyson Molekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-ecdyson-molekul-47691607.html
Ecdyson Molekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-ecdyson-molekul-47691607.htmlRFCNGF3K–Ecdyson Molekül
 Vitamin B6 (Pyridoxal) Molekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-vitamin-b6-pyridoxal-molekul-47692657.html
Vitamin B6 (Pyridoxal) Molekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-vitamin-b6-pyridoxal-molekul-47692657.htmlRFCNGGD5–Vitamin B6 (Pyridoxal) Molekül
 Carisoprodol Wirkstoffmolekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-carisoprodol-wirkstoffmolekul-47691323.html
Carisoprodol Wirkstoffmolekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-carisoprodol-wirkstoffmolekul-47691323.htmlRFCNGENF–Carisoprodol Wirkstoffmolekül
 Vitamin B12 (Cobalamin)-Molekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-vitamin-b12-cobalamin-molekul-47692645.html
Vitamin B12 (Cobalamin)-Molekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-vitamin-b12-cobalamin-molekul-47692645.htmlRFCNGGCN–Vitamin B12 (Cobalamin)-Molekül
 Trichloressigsäure-Molekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-trichloressigsaure-molekul-47692570.html
Trichloressigsäure-Molekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-trichloressigsaure-molekul-47692570.htmlRFCNGGA2–Trichloressigsäure-Molekül
 Orlistat Wirkstoffmolekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-orlistat-wirkstoffmolekul-47692170.html
Orlistat Wirkstoffmolekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-orlistat-wirkstoffmolekul-47692170.htmlRFCNGFRP–Orlistat Wirkstoffmolekül
 Triclosan-Molekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-triclosan-molekul-47692571.html
Triclosan-Molekül Stockfotohttps://www.alamy.de/image-license-details/?v=1https://www.alamy.de/stockfoto-triclosan-molekul-47692571.htmlRFCNGGA3–Triclosan-Molekül

